前回の続きです。クレンジングしたデータを使って予測モデルを構築していきます。
予測モデル構築編の4回目は、「Uni-Mol」を用いたモデルを構築したいと思います。
Uni-Mol について
Uni-Molは、Transformerベースの事前学習モデルを活用した特徴量抽出と、それらを下流タスクに適用するためのファインチューニングで構成される表現学習フレームワークです。
分子やタンパク質の三次元情報を取り扱うよう設計されており、分子のプロパティ予測の他、コンフォメーション予測やタンパク質との複合体予測など汎用的に利用可能です。
Uni-Molは、次の3つのパートで構成されてます(以下、各パートについて説明を入れますが、論文の内容をざっくりとまとめたもので正確性に欠ける部分があることにご注意ください)。
- Backborn:骨格モデル
- Pretraining:事前学習
- Finetuning:ファインチューニング
骨格モデルは、三次元空間の特徴を直接取り扱うことができるように拡張したTransformerベースのモデルを利用しています。
事前学習では、「2.08億(208M)の分子コンフォメーション」と「320万(3.2M)のタンパク質ポケット」をデータセットとして使用し、自己教師あり学習として「3D位置復元(3D position recovery)」と「マスクされた原子予測(masked atom prediction)」の損失を最適化しています。
ファインチューニングでは、事前学習モデルから得られた 「原子表現(Atom representation)」 および 「ペア表現(Pair representation)」 を利用して下流タスクの予測を行います。
Uni-Molは2023年の登場以降、各種ケモインフォマティクス系のタスクで上位を維持しています。
内容的には難しいことをやっていますが、三次元情報とTransformerをかけ合わせたユニークな手法ということもあり、どのような特徴があるか早めに知っておいて損はないと思います。
モデル構築の流れ
モデル構築は以下の流れで行います。
- モデル構築と精度検証
モデル構築と精度検証
今回は、公式が提供する「Uni-Mol Tools」というプロパティ予測のためのラッパーを利用します。
Uni-Mol単体でもプロパティ予測は可能ですが、分子の座標生成や推論などの関数を自力で定義する必要がありかなり大変です。
一方、Uni-Mol Toolsでは、プロパティ予測に必要な関数が一通り提供されており、かつscikit-learn風のAPI設計となっているため非常に使いやすくなっています。
2025年3月現在、Uni-Mol Toolsはベータ版ということもあり、痒いところに手が届かない部分もまだまだ残っていますが、それでも圧倒的に便利なので今回はこちらを使っていきたいと思います。
実装
環境構築
事前学習モデルはサイズが大きいため、ローカルのPCではGPUメモリが不足するかもしれません。
そのため、今回はローカル用に加えてGoogle Colabo用の環境構築方法も紹介します。
ローカルのPCで必要なGPUメモリの目安は8GBです。管理人のGPUが8GBであり、Uni-Molの最小モデルがギリギリ動くことを確認しています。
ローカル用
gitコマンドを使用します。設定がまだの方はこちらのサイト等を参考に設定しましょう。
構築方法の選択肢が幅広いので、管理人が構築したときの依存性とパッケージ管理の方法を以下に記載します。conda環境です。
作業ディレクトリにUni-Molのフォルダをダウンロードするため、任意のディレクトリに移動しておきましょう。Pythonのバージョンは3.9でも問題ありません。
cd (任意のディレクトリ)
conda create -n UniMolTools python=3.8
conda activate UniMolTools
pip install torch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 --index-url https://download.pytorch.org/whl/cu118
pip install huggingface_hub
git clone https://github.com/deepmodeling/Uni-Mol.git
cd Uni-Mol/unimol_tools
pip install .
conda install matplotlib
conda install jupyter環境構築ができたら、jupyter notebookを起動して以下のコードを実行しましょう。
import os
# 変数
HOME = os.getenv('HOME')
BASE_DIR = HOME + '/shaeo-blog/pj-logd'
MODEL_DIR = BASE_DIR + '/model/Uni-Mol'
TRAIN_DATA_PATH = BASE_DIR + '/dataset/train.csv'
TEST_DATA_PATH = BASE_DIR + '/dataset/test.csv'
os.environ["PYTORCH_CUDA_ALLOC_CONF"] = "max_split_size_mb:128"
# ファイルの作成
os.makedirs(MODEL_DIR, exist_ok=True)Google Colabo用
Google Driveのマイドライブに「Google Colabo」という名前でフォルダを作成し、その下にこれまで利用してきた「pj-logd」フォルダをコピーしましょう。
次に、Google Colaboを開いてノートブックを新規作成しましょう。
これで準備は完了です。以下、ノートブック上にコピーして実行する内容になります。
ランタイムの再起動を求められた場合は、再起動した後にもう一度上からセルを実行し直してください。
# ディレクトリのマウント
from google.colab import drive
drive.mount('/content/drive')# minicondaのインストール
%env PYTHONPATH=
!wget https://repo.anaconda.com/miniconda/Miniconda3-py39_23.11.0-2-Linux-x86_64.sh -O miniconda.sh
!bash miniconda.sh -b -f -p /usr/local
!rm miniconda.sh
!conda --version
!python -V# パッケージのインストール
!pip install torch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 --index-url https://download.pytorch.org/whl/cu118
!pip install huggingface_hub
!pip install matplotlib-venn# Uni-Molのダウンロード
%cd drive/MyDrive/'Google Colab'/pj-logd
!git clone https://github.com/deepmodeling/Uni-Mol.git
%cd Uni-Mol/unimol_tools
!pip install .
%cd ../../# 各種設定
import os, sys
# 変数
BASE_DIR = '/content/drive/MyDrive/Google Colab/pj-logd'
UNIMOL_TOOLS_DIR = BASE_DIR + '/Uni-Mol/unimol_tools'
MODEL_DIR = BASE_DIR + '/model/Uni-Mol'
TRAIN_DATA_PATH = BASE_DIR + '/dataset/train.csv'
TEST_DATA_PATH = BASE_DIR + '/dataset/test.csv'
os.environ["PYTORCH_CUDA_ALLOC_CONF"] = "max_split_size_mb:128"
# ファイルの作成
os.makedirs(MODEL_DIR, exist_ok=True)
# パスの追加
sys.path.append('/usr/local/lib/python3.9/site-packages')上記は今回の検証で一応動いた環境の構築方法です。こちらで意図していない動きを放置している箇所もあるため、変更して利用する場合などはご注意ください。
モデル構築と精度検証
以下のコードを実行してモデルを構築します。
Uni-Mol Toolsでは、事前学習モデルのダウンロード、データの前処理、ファインチューニング、モデルの保存、といった流れがシームレスに行われます。
また、k分割交差検証法がデフォルトで設定されており、スタッキングモデルへ組み込むことも容易です。
今回はスタッキングを利用しないため気にする必要はありませんが、訓練データの予測結果は”pred”ではなく”model.cv_pred”に保存されるため注意しましょう。
座標の生成と訓練を合わせると結構な時間がかかります。管理人の環境で2時間程度でした。
# モデルの設定
from unimol_tools import MolTrain
model = MolTrain(
task='regression', # タスクの種類
data_type='molecule', # インプットの種類
epochs=30, # ファインチューニングのエポック数
learning_rate=1e-4, # 学習率
batch_size=8, # バッチサイズ(8より大きいと8GBのGPUでは落ちる)
early_stopping=5, # Early Stopping
split='random', # 交差検証法の分割方法
kfold=5, # 交差検証法の分割数
save_path=MODEL_DIR, # モデルの保存先
smiles_col='canonical_smiles', # SMILESの列名
target_cols='standard_value', # タスクの列名
use_cuda=True, # GPUを利用するかどうか
use_amp=True, # AMP(Automatic Mixed Precision)を利用するかどうか
load_model_dir=None, # 事前学習モデルのロード先
model_name='unimolv1', # 利用するUni-Molのバージョン
model_size='84m', # 利用するモデルのサイズ
)
# 学習
pred = model.fit(data=TRAIN_DATA_PATH) # predの返り値はNone。予測結果はmodel.cd_pred訓練が完了したら、テストデータの予測に移る前に訓練に利用したモデルを削除してGPUメモリを開放します。
Uni-Mol Toolsでは、訓練とは別に予測用のモデルをインスタンス化する必要があるため、訓練で利用したモデルは削除して問題ありません。
# GPUメモリの開放
import torch
del model # 変数の削除
torch.cuda.empty_cache() # 空きメモリを増やすでは、以下のコードを実行してテストデータを予測しましょう。Uni-Mol Toolsでは、交差検証法で訓練された各モデルの予測値の相加平均が最終的な予測値となります。
予測値の算出も座標の生成が律速で時間がかかります。1時間程度気長に待ちましょう。
# テストデータの予測
model_eval = MolPredict(load_model=MODEL_DIR)
pred_test = model_eval.predict(data=TEST_DATA_PATH)最後に散布図を作成して予測精度を確認してみましょう。
# 散布図(テストデータ)
import pandas as pd
from sklearn.metrics import r2_score
import matplotlib.pyplot as plt
# データの読み込み
df_test = pd.read_csv(TEST_DATA_PATH)
# 予測値の算出
r2_test = r2_score(df_test['standard_value'], pred_test)
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(df_test['standard_value'], pred_test, label=f'Uni-Mol FT: R2:{r2_test:.3f}', alpha=0.5)
ax.legend()
plt.show()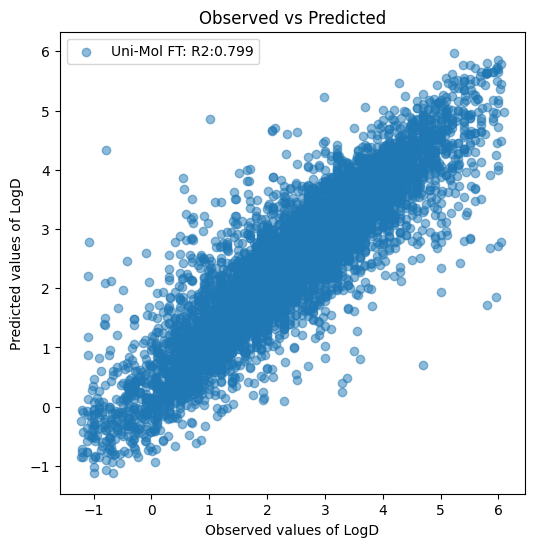
今までで一番R2が良かったAttentive FPの 0.738 を余裕で超えてますね。予測値の算出方法がこれまでと違うのでずるい気もしますが、そこは気にしないこととしましょう。
惜しくも R2 > 0.8 とはなりませんでしたが、流石はUni-Molです。
最後に
今回は、Uni-Molを用いてモデルを構築する方法を紹介しました。
Uni-Molはユニークな表現学習を行っているため、記述子やGNNとの相補性が高い(はず)です。
計算リソース的に全てのタスクでUni-Molを試すのが難しい場合は、いざというときのために一部のタスクでスタッキングの候補として使うところから始めてみるのも良いかもしれません。
また、今回を持ちましてPJ-LogDの本編は終了したいと思います。今後トピックを思いついた場合は、番外編という形で追加する予定です。
実務では既にツールが導入されており、モデル構築よりも予測値や寄与度を使った考察・意思決定の方が重視されるかもしれません。
しかしながら、ツールのベースとなっている技術に理解がない状態では、質の高い考察や意思決定は生まれないと個人的には感じています。
PJ-LogDは、ケモインフォマティクス初学者〜中級者向けの内容ですが、日本語での情報収集が難しい部分をカバーしているつもりです。そのため、既存の情報と相補的に利用していただけると幸いに思います。





コメント