前回の続きです。クレンジングしたデータを使って予測モデルを構築していきます。
予測モデル構築編の3回目は、特徴量としてRDkit記述子、アルゴリズムとして「TabNet」を用いたモデルを構築していきます。
TabNet について
TabNetは、表形式データ(Tabular data)の学習に特化したニューラルネットワークモデルです。
表形式のデータは従来型の機械学習(特に決定木系)の方が向いているタスクが多いですが、TabNetはそれに匹敵する、あるいは超える性能をもつモデルだと言われています。
特徴は以下の通りです。元論文のイントロに記載されている内容を参考にしています。文末のカッコは管理人の解釈です。
- 前処理が不要(特徴量エンジニアリングが不要)
- エンドツーエンドの学習に柔軟に統合することが可能(マルチモーダルモデルへの統合が容易)
- 注意機構(Attention)を用いたマスクによる特徴量選択と解釈性の向上
- 教師なし事前学習(Unspervised pre-training)が可能
今回は深堀しませんが、解釈性は”局所的な解釈”と”広域的な解釈”の双方に対応しています。
他のモデルより圧倒的に優れているわけではないのであまり注目されていない印象ですが、個人的にはかなり面白いモデルだと思っています。
モデル構築の流れ
モデル構築は以下の流れで行います。
- 特徴量の算出
- モデル構築と精度検証
特徴量の算出
今回は「RDkit記述子」を利用します。
記述子(Descriptor)は”データの特徴を数値化・定量化して表現する指標”のことであり、化合物の記述子としては以下のようなものが挙げられます。
- 分子量
- 水素結合供与体数・水素結合受容体数
- トポロジカル極性表面積(TPSA)
RDkitでは化合物の基本的な記述子が算出可能であり、今回はその中でも0D~2D情報に限定した記述子を利用したいと思います。
ちなみに、広義の意味ではフィンガープリントも化合物の記述子に含まれますが、ケモインフォマティクスでは”記述子”と”フィンガープリント”を別物として扱うことが多いです。
モデル構築と精度検証
TabNetでは、教師なし事前学習が可能です。
TabNetの教師なし事前学習は、「自己教師あり学習(self-supervised learning)」の一種として設計されており、ランダムにマスクした特徴量を予測し、その誤差が最小となるようにモデルを訓練します。
元論文では教師なし事前学習による大幅なパフォーマンス向上が認められているため、今回のモデル構築でも比較して見たいと思います。
また、今回は特徴量として RDkit Descriptor を利用します。これまで構築したモデルの中にRDkit Descriptor を使ったものはないため、TabNetモデルの精度に違いがあっても特徴量の影響下アルゴリズムの影響か特定するのが難しいです。
そこで最後はランダムフォレストモデルを構築し、従来型の機械学習とTabNetにどの程度精度の違いがあるかについても確認してみたいと思います。
実装
環境構築
論文をもとに実装された「pytorch-tabnet」を利用します。構築方法の選択肢が幅広いので、管理人が構築したときの依存性とパッケージ管理の方法を以下に記載します。conda環境です。
- python 3.8
- pytorch-tabnet 4.1.0 (pip: default)
- RDkit 2024.3.5 (pip: default)
- pandas 2.0.3 (pip: default)
- jupyter 1.1.1 (conda: default)
- matplotlib 3.7.3 (conda: default)
特徴量の算出
RDkitで算出可能な記述子は以下のモジュールで確認できます。
- rdkit.Chem.Descriptors
- rdkit.Chem.Descriptors3D
今回は2D情報までの記述子を利用するため、1番目のDescriptorsモジュールで定義されている210個の記述子を利用します。
210個の記述子はだいたいの化合物で計算できますが、一部の化合物(チャージを持つ、分子量が大きすぎる、など)で計算エラーとなるものも存在しています。
今回のデータは程々のクレンジングしかしていないので、デフォルト設定で記述子を計算すると欠損値が発生してしまいます。
まずはそれらを計算対象から外すために、以下のコードで実際に利用する記述子をリストとして取得しましょう。
from rdkit.Chem import Descriptors
# すべての記述子のリストを取得
descriptor_dict = dict(Descriptors.descList)
descriptor_names = descriptor_dict.keys()
# 一覧を表示
print('RDkitで計算可能な記述子の数(3D記述子を除く): ', len(descriptor_names))
print(descriptor_names)
# エラーが発生しやすい記述子を削除する
selected_descriptor_names = []
for desc_name in descriptor_names:
if not (desc_name=='Ipc' or 'BCUT2D' in desc_name or 'Charge' in desc_name):
selected_descriptor_names.append(desc_name)
print('予測モデル構築に利用する記述子の数: ', len(selected_descriptor_names))
print(selected_descriptor_names)計算エラーが発生しやすいものを除くと197個の記述子が残りました。
では、実際に記述子を計算していきましょう。記述子の計算は以下のクラスを利用すると便利です。
- rdkit.ML.Descriptors.MoleculeDescriptors.MolecularDescriptorCalculator
使い方は以下の通りです。化合物ごとに記述子を計算し、結果をタプルとして取得しています。また、訓練・テストデータそれぞれでリストを作成し計算結果を格納しています。
import pandas as pd
from rdkit import RDLogger, Chem
from rdkit.ML.Descriptors.MoleculeDescriptors import MolecularDescriptorCalculator
# データの読み込み
df_train = pd.read_csv('~/shaeo-blog/pj-logd/dataset/train.csv')
df_test = pd.read_csv('~/shaeo-blog/pj-logd/dataset/test.csv')
# ログレベルを調整する
lg = RDLogger.logger()
lg.setLevel(RDLogger.CRITICAL)
# 記述子の計算
calculator = MolecularDescriptorCalculator(selected_descriptor_names)
descriptors_train = []
descriptors_test = []
for smi_train, smi_test in zip(df_train['canonical_smiles'], df_test['canonical_smiles']):
mol_train = Chem.MolFromSmiles(smi_train)
descriptors_train.append(calculator.CalcDescriptors(mol_train))
mol_test = Chem.MolFromSmiles(smi_test)
descriptors_test.append(calculator.CalcDescriptors(mol_test))少し時間がかかりますが、これで記述子を計算することができました。
後ほど使いやすいようにデータをpandasのDataFrame型に変換しておきましょう。また、次回以降も利用できるようにバイナリ型としてデータを保存しておきましょう。
import os
import pickle
# モデル用のデータ設定
y_train = df_train['standard_value']
y_test = df_test['standard_value']
X_train = pd.DataFrame(data=descriptors_train, columns=calculator.GetDescriptorNames(), index=df_train.index)
X_test = pd.DataFrame(data=descriptors_test, columns=calculator.GetDescriptorNames(), index=df_test.index)
# データ保存先のパス設定
HOME = os.getenv('HOME')
DATASET_DIR = HOME + '/shaeo-blog/pj-logd/dataset'
# 特徴量を保存する
with open(os.path.join(DATASET_DIR, 'X_train_rddesc.pkl'), mode='wb') as f:
pickle.dump(X_train, f)
with open(os.path.join(DATASET_DIR, 'X_test_rddesc.pkl'), mode='wb') as f:
pickle.dump(X_test, f)モデル構築と精度検証
データの設定
モデルの学習用に特徴量の正規化(min-maxスケーリング)を行います。
本来、TabNetのアーキテクチャを考えると特徴量の正規化は必要ありませんが、「pytorch-tabnet」の事前学習は平均二乗誤差(MSE)で最適化を行っているため特徴量のスケーリングが必要です。
今回は事前学習あり・なしの双方を検討するため、はじめから特徴量を正規化しておきます。
また、「pytorch-tabnet」はEarly Stoppingが容易に実装できるため検証データを作成します。
実装は以下の通りです。
from sklearn.preprocessing import MinMaxScaler
# 正規化(min-max)
scaler = MinMaxScaler()
scaler.fit(X_train)
X_train = pd.DataFrame(data=scaler.transform(X_train), index=X_train.index, columns=X_train.columns)
X_test = pd.DataFrame(data=scaler.transform(X_test), index=X_test.index, columns=X_test.columns)
# 検証用データの設定
y_valid = y_train.sample(frac=0.2, random_state=0)
y_train = y_train.drop(index=y_valid.index)
X_valid = X_train.iloc[y_valid.index, :]
X_train = X_train.drop(index=X_valid.index)事前学習なし
「pytorch-tabnet」は、sklearn-likeなAPIで実装されているため容易にモデルを構築することができます。
まずはモデルの設定を行います。実装は以下の通りです。
インスタンス化の設定は基本デフォルトの値ですが、”n_d”と”n_a”はデフォルトの値である「8」から「128」に変更しています。
from pytorch_tabnet.tab_model import TabNetRegressor
# Fine-tuningモデル
model = TabNetRegressor(
n_d=128, # 出力層の次元数
n_a=128, # Attention層の次元数
n_steps=3, # ネットワークのステップ数
gamma=1.3, # Attentionを更新するときにスケーリングファクター
n_independent=2, # 各GLUの層数?
n_shared=2, # 各GLUの層数?
epsilon=1e-15, # Log(0) を防ぐため
momentum=0.02, # バッチ正規化のモーメンタム
lambda_sparse=1e-3, # スパース変換で生じた誤差の補正項
seed=0, # 初期シード
clip_value=1, # 勾配爆発を防ぐための正則化項
verbose=1, # 訓練時の出力?
mask_type="sparsemax", # maskの手法
input_dim=None, # 入力の特徴量次元数(自動)
output_dim=None, # 出力の特徴量次元数(自動)
device_name="auto", # 使用するデバイス(自動)
)次にモデルの訓練を行います。実装は以下の通りです。
こちらも基本的にデフォルトの値でから変更していませんが、”max_epochs”と”patience”は大きめの値を設定しています。
# データをndarrayに変換する
X_train = X_train.values
y_train = y_train.values.reshape(-1, 1)
X_valid = X_valid.values
y_valid = y_valid.values.reshape(-1, 1)
X_test = X_test.values
y_test = y_test.values.reshape(-1, 1)
# モデルの訓練
model.fit(
X_train,
y_train,
eval_set=[(X_train, y_train), (X_valid, y_valid)],
eval_name=['train', 'valid'],
eval_metric=['mae', 'rmse', 'mse'],
max_epochs=1000,
patience=100,
batch_size=1024,
virtual_batch_size=128,
num_workers=0,
drop_last=True,
compute_importance=True # 特徴量の重要度を計算するかどうか
)訓練が終了したら以下のコードで学習曲線を作成して確認してみましょう。
import matplotlib.pyplot as plt
epochs = [i+1 for i in range(len(model.history['train_mse']))]
# 学習曲線の描画
plt.plot(epochs, model.history['train_mse'], linestyle='-', color='b', label='Train MSE')
plt.plot(epochs, model.history['valid_mse'], linestyle='-', color='r', label='valid MSE')
# ラベルとタイトルの設定
plt.xlabel('Epochs')
plt.ylabel('MSE')
plt.title('Learning Curve (MSE over Epochs)')
plt.legend()
# グラフを表示
plt.show()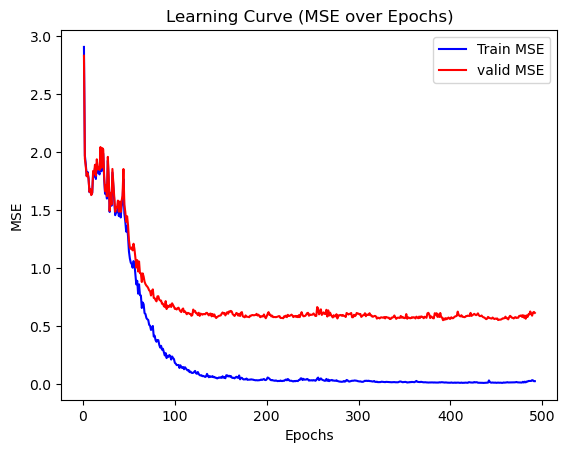
〜50エポックあたりでは学習が不安定ですが、後半になるにつれて安定しているので収束していると考えても良さそうですね。
学習に使っていないテストデータで評価値(R2)を算出し、モデルがどの程度の精度になったか確認してみましょう。
# モデルの評価
from sklearn.metrics import r2_score
import matplotlib.pyplot as plt
# テストデータの予測値を算出する
y_pred_test = model.predict(X_test)
r2_test = r2_score(y_test, y_pred_test)
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(y_test, y_pred_test, label=f'TabNet RDkit desc: R2:{r2_test:.3f}', alpha=0.5)
ax.legend()
plt.show()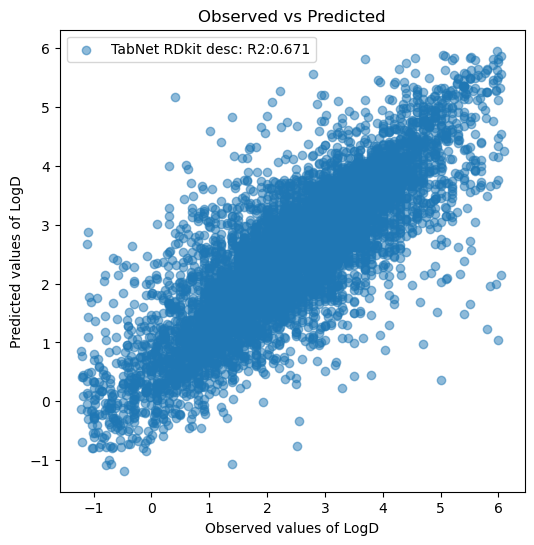
前回のAttentive FPモデル(R2:0.738)より劣るものの、そこそこの精度が出ていますね。
事前学習あり
事前学習を行うことで精度が改善されるか試してみたいと思います。
TabNetの事前学習はエンコーダーデコーダーを利用しており、学習したエンコーダーのパラメータをFine Tuningし推論に利用するようです。
まずはモデルを設定します。実装は以下の通りです。
from pytorch_tabnet.pretraining import TabNetPretrainer
# 事前学習モデル
unsupervised_model = TabNetPretrainer(
n_d=128, # 出力層の次元数
n_a=128, # Attention層の次元数
seed=0, # 初期シード,
optimizer_params=dict(lr=2e-2),
mask_type='entmax' # "sparsemax"
)
# Fine-tuningモデル
model_ft = TabNetRegressor(
n_d=128, # 出力層の次元数
n_a=128, # Attention層の次元数
seed=0, # 初期シード
verbose=1, # 訓練時の出力?
mask_type="sparsemax", # maskの手法
device_name="auto", # 使用するデバイス(自動)
)次にモデルの訓練を行います。実装は以下の通りです。
# 自己教師あり
unsupervised_model.fit(
X_train=X_train,
eval_set=[X_valid],
max_epochs=1000,
patience=100,
#pretraining_ratio=0.5,
)
# モデルの訓練
model_ft.fit(
X_train,
y_train,
eval_set=[(X_train, y_train), (X_valid, y_valid)],
eval_name=['train', 'valid'],
eval_metric=['mae', 'rmse', 'mse'],
max_epochs=1000,
patience=100,
batch_size=1024,
virtual_batch_size=128,
num_workers=0,
drop_last=True,
from_unsupervised=unsupervised_model,
)訓練が終了したら以下のコードで学習曲線を作成して確認してみましょう。
# 学習状況を描写する
import matplotlib.pyplot as plt
epochs = [i+1 for i in range(len(model_ft.history['train_mse']))]
# 学習曲線の描画
plt.plot(epochs, model_ft.history['train_mse'], linestyle='-', color='b', label='Train MSE')
plt.plot(epochs, model_ft.history['valid_mse'], linestyle='-', color='r', label='valid MSE')
# ラベルとタイトルの設定
plt.xlabel('Epochs')
plt.ylabel('MSE')
plt.title('Learning Curve (MSE over Epochs)')
plt.legend()
# グラフを表示
plt.show()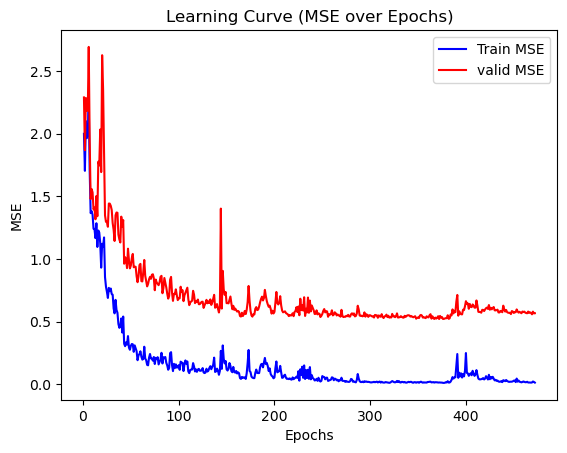
事前学習なしのときよりも学習が安定していないですね。Early Stoppingが早すぎたかもしれませんが、最終的には一定の値に落ち着いているので収束していると考えましょう。
モデルがどの程度の精度になったか確認してみましょう。
# テストデータの予測値を算出する
y_pred_ft_test = model_ft.predict(X_test)
r2_test_ft = r2_score(y_test, y_pred_ft_test)
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(y_test, y_pred_ft_test, label=f'TabNet-FT RDkit desc: R2:{r2_test_ft:.3f}', alpha=0.5)
ax.legend()
plt.show()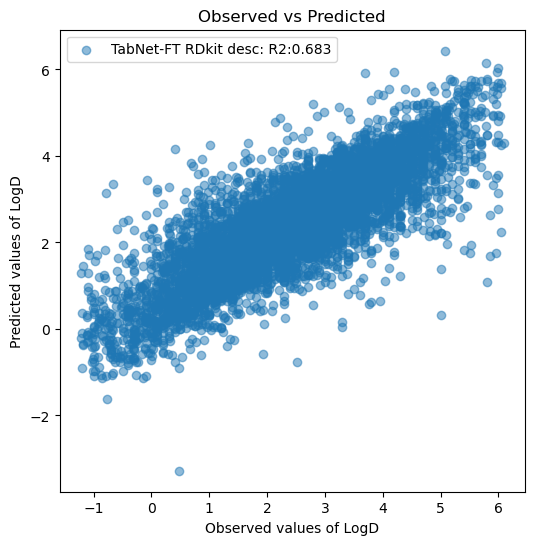
事前学習なしの場合のR2が0.671なので、数値上は精度が上がってますね。ただし、誤差が大きいサンプルが目立っているので、今回のケースについては事前学習をした方が良いかは微妙なところです。
作成したモデルは以下のコードで保存しておきましょう
# モデルを保存する
model_path = HOME + '/shaeo-blog/pj-logd/model/TabNet_rddesc'
model_ft_path = HOME + '/shaeo-blog/pj-logd/model/TabNet_FT_rddesc'
model.save_model(model_path)
model_ft.save_model(model_ft_path)ランダムフォレストモデル(比較用)
TabNetがアルゴリズムとして強力かどうかを確かめるため、ランダムフォレストと比較してみたいと思います。
実装は以下の通りです。ハイパーパラメータはデフォルトのままモデルを構築します。
import numpy as np
from sklearn.ensemble import RandomForestRegressor
model_rf = RandomForestRegressor(n_jobs=-1, random_state=0)
model_rf.fit(np.concatenate((X_train, X_valid), axis=0), np.concatenate((y_train, y_valid), axis=0))
y_pred_rf_test = model_rf.predict(X_test)
r2_test_rf = r2_score(y_test, y_pred_rf_test)
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(y_test, y_pred_rf_test, label=f'RF RDkit desc: R2:{r2_test_rf:.3f}', alpha=0.5)
ax.legend()
plt.show()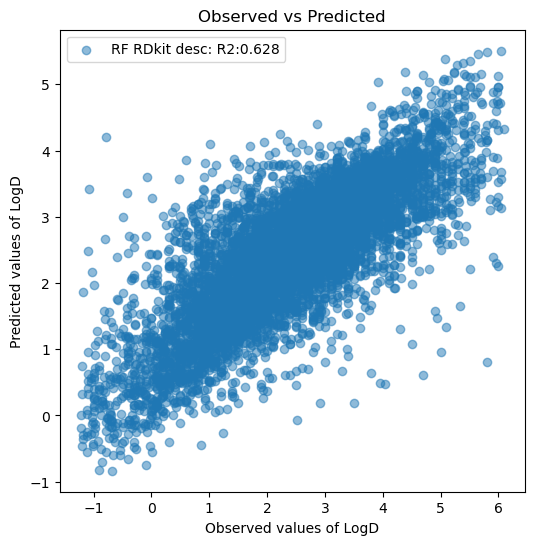
R2は、事前学習なしTabNetが0.671、事前学習ありのTabNetが0.683でした。
勾配ブースティング決定木など他のアルゴリズムやハイパーパラメータの最適化を考える必要はあるものの、RDkit Descriptorsを用いたLogD予測に関しては従来型よりTabNetの方が強力なのかもしれません。
今回は触れませんが、この辺りの考察は解釈性を深堀していくと面白いかもしれませんね。
最後に
今回は、RDkit記述子とTabNetを用いてモデルを構築する方法を紹介しました。
今回のケースではTabNetが有用な手法だと考えられる結果が得られましたが、学習が安定しなかったり、高次元のデータに対応しきれなかったり、実はかなりクセがある手法であることには注意が必要です(Morgan Fingerprintで試したときは学習が上手く行かずR2が0を下回りました)。
次回のモデル構築編4回目は、Uni-Molというフレームワークを試したいと思います。引き続きご覧いただけますと幸いです。





コメント