前回の続きです。クレンジングしたデータを使って予測モデルを構築していきます。
予測モデル構築編の1回目は、特徴量としてMorgan Fingerprint、アルゴリズムとして勾配ブースティング系を用いて高精度なモデルを目指していこうと思います。
モデル構築の流れ
今回のモデル構築は以下の流れで行います。
- 特徴量の算出
- モデル構築と精度検証
- スタッキングモデルの構築と精度検証
特徴量の算出
特徴量は「Morgan Fingerprint」を使用します。
Morgan Fingerprint は、分子のグラフ表現をもとに算出される特徴量であり、特定の原子を中心に半径(radius)内の環境を考慮し、原子や結合の情報をハッシュ関数を用いて固定長のビットベクトルに変換する方法です。
化合物のプロパティ予測モデルの特徴量としては分子記述子や分散表現などもありますが、計算が安定していて扱いやすいことから今回はこちらのみを利用していきたいと思います。
また、Morgan Fingerprint は半径や ビット長、グラフ表現のノードやエッジ(原子や結合)に与える情報によっても特徴量としての性質が変わってきます。
色々試すのが望ましいですが、全てを試していてはキリがないため今回は「半径2、1,024ビット(次元)、ECFP相当」の条件で利用します。
モデル構築と精度検証
勾配ブースティング決定木(Gradient Boosting Tree)系のアルゴリズム3種、XGBoost、LightGBM、CatBoostでモデルを構築します。
勾配ブースティング決定木とは、1つ前のモデルの予測誤差が小さくなるように学習していくアンサンブル学習「ブースティング」をベースとしたモデルであり、アンサンブルに利用するアルゴリズムに「決定木」、誤差の学習に損失関数の「勾配(gradient)」を用いているためこのように呼ばれています。
ディープラーニングが流行している現在も表(テーブル)データに対して非常に有効な手法として用いられています。各ベンチマークにおいても従来型の機械学習の中でのトップはだいたい勾配ブースティング系です。
XGBoost、LightGBM、CatBoostは、勾配ブースティング決定木の実装です。年代順に並んでいていますがタスクによって最も精度が良いものが変わってくるので3つ全てを試したいと思います。
また、予測モデルを構築する際はハイパーパラメータが重要となりますが、上記のアルゴリズムではハイパーパラメータの影響が(比較的)小さいため今回は最適化を行いません。
スタッキングモデルの構築と精度検証
「スタッキング」は「ブースティング」や「バギング」とならぶアンサンブル学習の手法の1つです。
スタッキングでは、線型性の特徴と捉える重回帰分析、非線形の関係を捉える決定木・・・のように性質が異なる複数のモデルの予測値を「メタ特徴量」として、データセットの特徴を余すことなく利用します。
スタッキングが効果的な手法かどうかはイメージしづらいと思いますが、ディープラーニングの表現学習を複数のモデルで代替していると考えればしっくりくるのではないでしょうか?
テーブルデータや少数データの場合は表現学習が不十分なことが多いため、ディープラーニング(単体)よりもスタッキングモデルの方が強い多い印象です。
前項の3モデルは勾配ブースティング決定木に分類されますが、それぞれ特徴が異なるためスタッキングによる精度向上が期待できます。
スタッキングすれば必ずしも精度が上がるというわけではないですし解釈性が下がるという懸念もありますが、ものは試しということでスタッキングの効果も確認したいと思います。
実装
前回に加えて以下のPythonライブラリを新たに利用します。仮想環境にpipまたはcondaでインストールしましょう(pip install xxx あるいは conda instal xxx、xxxはライブラリ名)。
- xgboost
- lightgbm
- catboost
- matplotlib
特徴量の算出
RDkitでMorgan Fingerprintを算出します。以下のコードを実行してください。RDkitは古いバージョンだとrdFingerprintGeneratorが実装されていない可能性があるのでご注意ください。
import numpy as np
import pandas as pd
from rdkit import Chem
from rdkit.Chem import rdFingerprintGenerator
# Morgan Fingerprintを計算する関数
mfpgen = rdFingerprintGenerator.GetMorganGenerator(radius=2,fpSize=1024) # 半径=2, ビット長=2048, ECFP相当
def calc_mfingerprint(df, smi_col='canonical_smiles'):
list_fp = []
s_mol = df[smi_col].map(Chem.MolFromSmiles) # RDkitのMoloオブジェクトを生成する
for mol in s_mol:
fp = mfpgen.GetFingerprint(mol) # Morgan Fingerprintを算出(ExplicitBitVectオブジェクト)
list_fp.append(np.array(fp)) # numpyのndarray型に変換してリストに追加
return list_fp # 化合物のMorgan Fringerprintを要素に持つリストを返す
# データの読み込み
df_train = pd.read_csv('~/shaeo-blog/pj-logd/dataset/train.csv')
df_test = pd.read_csv('~/shaeo-blog/pj-logd/dataset/test.csv')
# Morgan Fringerprintを算出
list_fp_train = calc_mfingerprint(df_train)
list_fp_test = calc_mfingerprint(df_test)訓練データとテストデータそれぞれに対して、1,024次元の特徴量(numpyのndarray型)を要素に持つリストが作成されました。
この状態ではモデルにインプットすることができないので、以下のコードで目的変数と特徴量ともにモデルへインプットできる状態にしましょう。
特徴量の計算はそこそこ時間がかかるのでバイナリファイルとしてキャッシュしておきます。
import os
from pathlib import Path
import pickle as pkl
# データの設定
y_train, y_test = df_train['standard_value'], df_test['standard_value'] # 目的変数
X_train, X_test = pd.DataFrame(list_fp_train), pd.DataFrame(list_fp_test) # 説明変数(特徴量)
df_train_pc = pd.concat([y_train, X_train], axis=1)
df_test_pc = pd.concat([y_test, X_test], axis=1)
# 特徴量を保存する
DATASET_DIR = Path.home() / 'shaeo-blog/pj-logd/dataset'
with open(os.path.join(DATASET_DIR, 'X_train_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(X_train, f)
with open(os.path.join(DATASET_DIR, 'X_test_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(X_test, f)モデル構築と検証
XGBoost、LighGBM、CatBoost、それぞれのモデルを実装していきます。
Pythonでは機械学習モデルが簡単に実装できるようになっていますが、その理由の1つがscikit-leanのAPIが浸透していることにあります。
すなわち、だいたいのモデルを「モデルクラスのインスタンス化」と「訓練」の2ステップで作成することができるということです。上記3つのモデルに関しても同様であり、ローコードでモデルを作成することができます。
XGBoostモデル
以下、XGBoostを実装するコードです。モデルクラスをインスタンス化する際に各種設定ができます。
今回ハイパーパラメータはデフォルトで使用しますが、よく使うものはコメントアウトして記述しておきます(初期シードとCPUの使用コア数、メッセージの出力設定はいじります)。
from xgboost import XGBRegressor
# モデルクラスのインスタンス化
# model_xgb = XGBRegressor() でOK
model_xgb = XGBRegressor(
#max_depth=6, # 各決定木の最大の深さ
#max_leaves=0, # 各決定木のノードの数(0は無制限)
#grow_policy='depthwise', # 決定木の分割に利用する手法(LightGBM様の"lossguide"も実装可能)
#learning_rate=0.3, # 学習率
#n_estimators=100, # ブースティングに利用する決定木の数
verbosity=0, # 0 (silent) - 3 (debug)
#booster='gbtree', # 勾配ブースティングで用いるモデル("gbtree", "gblinear", "dart")
n_jobs=-1, # 利用するCPUの数(-1は利用可能なCPUを全て利用する)
#subsample=1., # 各決定木で利用するサンプルの割合
#sampling_method='uniform', # 'gradient_based'
#colsample_bytree=1., # 各決定木で利用される特徴量の割合
#colsample_bylevel=1., # 決定木の深さ単位で利用される特徴量の割合
#colsample_bynode=1., # ノード分割で利用される特徴量の割合
#reg_alpha=0., # L1正則化
#reg_lambda=1.0, # L2正則化
random_state=42, # 初期シード
device='cpu', # 使用するデバイス
)
# モデルの構築
model_xgb.fit(X_train, y_train)LighGBMモデル
以下、XGBoostを実装するコードです。XGBoostと同様によく使うハイパーパラメータはコメントアウトして記述しておきます。
from lightgbm import LGBMRegressor
# モデルクラスのインスタンス化
# model_lgbm = LGBMRegressor() でOK
model_lgbm = LGBMRegressor(
#boosting_type="gbdt", # 勾配ブースティングで用いるモデル("gbdt", "dart", "rf")
#num_leaves=31, # 各決定木のノードの数
#max_depth=-1, # 各決定木の深さ(-1は無制限)
#learning_rate=0.1, # 学習率
#n_estimators=100, # ブースティングに利用する決定木の数
verbosity=-1, # 0 (silent) - 3 (debug)
n_jobs=-1, # 利用するCPUの数(-1は利用可能なCPUを全て利用する)
#subsample=1., # 各決定木で利用するサンプルの割合
#colsample_bytree=1., # 各決定木で利用される特徴量の割合
#reg_alpha=0., # L1正則化
#reg_lambda=1.0, # L2正則化
random_state=42, # 初期シード
device='cpu', # 使用するデバイス
)
# モデルの構築
model_lgbm.fit(X_train, y_train)CatBoostモデル
以下、CatBoostを実装するコードです。管理人はCatBoostをほぼ使わないのでハイパーパラメータ最適化の感覚はありませんが、使いそうなものはコメントアウトして記述しておきます。
from catboost import CatBoostRegressor
# モデルクラスのインスタンス化
# model_cb = CatBoostRegressor() でOK
model_cb = CatBoostRegressor(
#max_leaves=31, # 各決定木のノードの数
#depth=6, # 各決定木の深さ(CPUは~16, GPUは=8)
#learning_rate=0.03, # 学習率
#iterations=1000, # ブースティングに利用する決定木の数
verbose=0, # 0 (silent) - 3 (debug)
#subsample=1., # 各決定木で利用するサンプルの割合
#rsm=1., # 各決定木で利用される特徴量の割合
#l2_leaf_reg=1.0, # L2正則化
random_state=42, # 初期シード
devices='cpu', # 使用するデバイス
)
# モデルの構築
model_cb.fit(X_train, y_train)予測値の算出と精度の確認
3種のモデルの予測値を算出して決定係数(R2)を比較しましょう。また、実測値-予測値プロットを用いて相関を可視化してみましょう。
from sklearn.metrics import r2_score
import matplotlib.pyplot as plt
# 予測値の算出: XGBoost
y_test_pred_xgb = model_xgb.predict(X_test)
y_test_pred_xgb = pd.Series(data=y_test_pred_xgb, index=y_test.index, name='pred_xgb')
r2_score_xgb = r2_score(y_test, y_test_pred_xgb)
print('R2 XGBoost: ', f'{r2_score_xgb:.3f}')
# 予測値の算出: LightGBM
y_test_pred_lgbm = model_lgbm.predict(X_test)
y_test_pred_lgbm = pd.Series(data=y_test_pred_lgbm, index=y_test.index, name='pred_lgbm')
r2_score_lgbm = r2_score(y_test, y_test_pred_lgbm)
print('R2 LightGBM: ', f'{r2_score_lgbm:.3f}')
# 予測値の算出: CatBoost
y_test_pred_cb = model_cb.predict(X_test)
y_test_pred_cb = pd.Series(data=y_test_pred_cb, index=y_test.index, name='pred_cb')
r2_score_cb = r2_score(y_test, y_test_pred_cb)
print('R2 CatBoost: ', f'{r2_score_cb:.3f}')
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(y_test, y_test_pred_xgb, label=f'XGBoost: R2:{r2_score_xgb:.3f}', alpha=0.5)
ax.scatter(y_test, y_test_pred_lgbm, label=f'LightGBM: R2:{r2_score_lgbm:.3f}', alpha=0.5)
ax.scatter(y_test, y_test_pred_cb, label=f'CatBoost: R2:{r2_score_cb:.3f}', alpha=0.5)
ax.legend()
plt.show()
R2 XGBoost: 0.608
R2 LightGBM: 0.588
R2 CatBoost: 0.611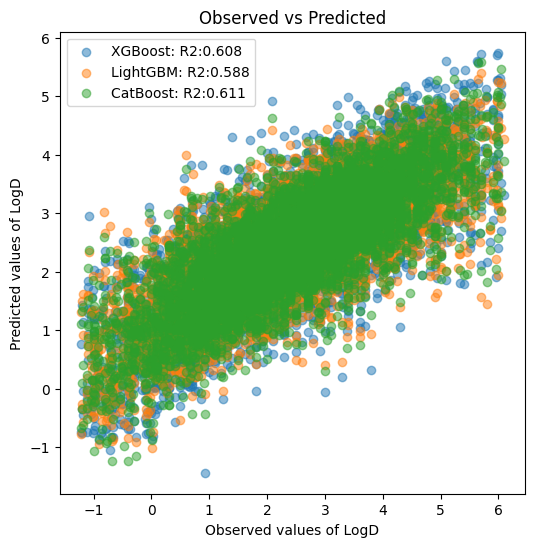
予測精度が高い順(決定係数が大きい順)に、CatBoost > XGBoost > LightGBM となりました。
決定係数が0.6以上なのでモデルとしてはそこそこの性能だと解釈できますが、LogDは分配係数なので「1の違いで脂溶性が10倍変わる」と思うとあまり使えそうにはありませんね…。そこまで高級な試験でもないため0.8以上の予測精度は欲しいですね。
特徴量がMorgan Fingerprintでハイパーパラメータがデフォルトならこんなものでしょう。
スタッキングモデルの構築と精度検証
XGBoost、LightGBM、CatBoost、これをベースモデルとしてスタッキングモデルを構築します。
スタッキングモデルでは、テストデータのメタ特徴量として”全訓練データで構築したベースモデルの予測値”、訓練データのメタ特徴量として”k分割交差検証法(k-fold cross-validation)の予測値”を利用します(そうしないとデータリーケージとなるためです)。
テストデータのメタ特徴量は前項で求めた予測値を利用できますが、訓練データは新たに交差検証法で予測値を算出する必要があります。
以下のコードで、5-fold cross-validationによる訓練データのメタ特徴量を算出とテストデータの予測値集約を行いましょう。
# 訓練データのメタ特徴量を算出する
from sklearn.model_selection import KFold
# クロスバリデーションの設定
kf = KFold(n_splits=5, shuffle=True, random_state=42)
# モデルの設定
model_xgb_cv = XGBRegressor(verbosity=0, n_jobs=-1, random_state=42, device='cpu')
model_lgbm_cv = LGBMRegressor(verbosity=-1, n_jobs=-1, random_state=42, device='cpu')
model_cb_cv = CatBoostRegressor(verbose=0, random_state=42, devices='cpu')
# 5-fold cross validation
X_train_meta = pd.DataFrame() # 保存用のデータフレーム
for i, (train_id_cv, valid_id_cv) in enumerate(kf.split(df_train)):
# データの設定
X_train_cv, X_valid_cv = X_train.iloc[train_id_cv], X_train.iloc[valid_id_cv]
y_train_cv, y_valid_cv = y_train.iloc[train_id_cv], y_train.iloc[valid_id_cv]
# モデルの訓練
model_xgb_cv.fit(X_train_cv, y_train_cv)
model_lgbm_cv.fit(X_train_cv, y_train_cv)
model_cb_cv.fit(X_train_cv, y_train_cv)
# 予測値の算出
y_valid_pred_xgb = model_xgb_cv.predict(X_valid_cv)
y_valid_pred_xgb = pd.Series(data=y_valid_pred_xgb, index=y_valid_cv.index, name='pred_xgb')
y_valid_pred_lgbm = model_lgbm_cv.predict(X_valid_cv)
y_valid_pred_lgbm = pd.Series(data=y_valid_pred_lgbm, index=y_valid_cv.index, name='pred_lgbm')
y_valid_pred_cb = model_cb_cv.predict(X_valid_cv)
y_valid_pred_cb = pd.Series(data=y_valid_pred_cb, index=y_valid_cv.index, name='pred_cb')
# 予測値の統合
df_valid_pred = pd.concat([y_valid_pred_xgb, y_valid_pred_lgbm, y_valid_pred_cb], axis=1)
X_train_meta = pd.concat([X_train_meta, df_valid_pred], axis=0)
# インデックスのソート
X_train_meta.sort_index(inplace=True)
# テストデータのメタ特徴量作成
X_test_meta = pd.concat([y_test_pred_xgb, y_test_pred_lgbm, y_test_pred_cb], axis=1)以上で訓練データとテストデータのメタ特徴量が用意できました。
これらを用いてスタッキングモデルを構築します。スタッキングに利用するアルゴリズムは何でもいいですが、Kaggle等のコンペで使用頻度の高い重回帰分析とXGBoostの2種でモデルを構築してみましょう。
# スタッキングモデルの作成
from sklearn.linear_model import LinearRegression
# 重回帰分析
stacker_mlr = LinearRegression()
stacker_mlr.fit(X_train_meta, y_train)
# XGBoost
stacker_xgb = XGBRegressor(verbosity=0, n_jobs=-1, random_state=42, device='cpu')
stacker_xgb.fit(X_train_meta, y_train)
# 予測値の算出: 重回帰分析
y_test_pred_s1 = stacker_mlr.predict(X_test)
y_test_pred_s1 = pd.Series(data=y_test_pred_s1, index=y_test.index, name='pred_stack_mrl')
r2_score_s1 = r2_score(y_test, y_test_pred_s1)
print('R2 stacking MLR: ', f'{r2_score_s1:.3f}')
# 予測値の算出: XGBoost
y_test_pred_s2 = stacker_xgb.predict(X_test)
y_test_pred_s2 = pd.Series(data=y_test_pred_s2, index=y_test.index, name='pred_stack_xgb')
r2_score_s2 = r2_score(y_test, y_test_pred_s2)
print('R2 stacking XGB: ', f'{r2_score_s2:.3f}')
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(y_test, y_test_pred_s1, label=f'stacking MLR: R2:{r2_score_xgb:.3f}', alpha=0.5)
ax.scatter(y_test, y_test_pred_s2, label=f'stacking XGB: R2:{r2_score_lgbm:.3f}', alpha=0.5)
ax.legend()
plt.show()R2 stacking MLR: 0.634
R2 stacking XGB: 0.579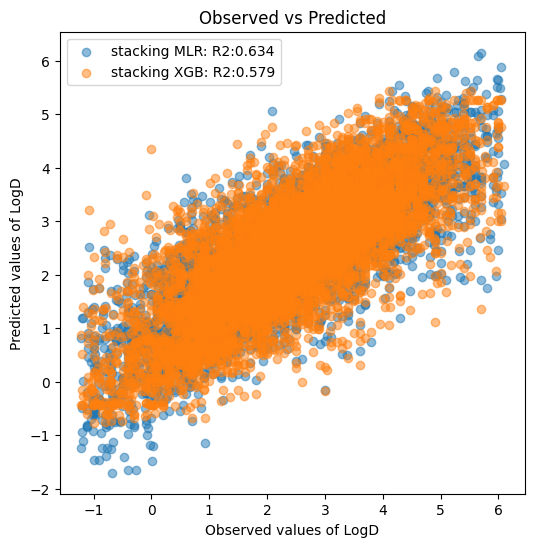
XGBoost、LightGBM、CatBoostの単独モデルと比較して、重回帰分析によるスタッキングではモデルの精度向上が認められました。
一方で、XGBoostによるスタッキングでは反対にモデルの精度が落ちました。
今回のようにベースモデルが少ないケースでは、スタッキングに用いるアルゴリズムとしてシンプルに線型性を捉える重回帰分析が強い印象です。XGBoostでのスタッキングは非線形の関係を捉えようとして失敗(過学習)してしまったのでしょうか?
最後に今回作成したモデルをバイナリファイルとして保存します。以降、バイナリファイルを読み込むことで訓練済みのモデルが使えるので予測したいデータがある場合は保存しておきましょう。
# モデルを保存する
MODEL_DIR = Path.home() / 'shaeo-blog/pj-logd/model'
os.makedirs(MODEL_DIR, exist_ok=True)
# XGBoost
with open(os.path.join(MODEL_DIR, 'XGBoost_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(model_xgb, f)
# LightGBM
with open(os.path.join(MODEL_DIR, 'LightGBM_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(model_lgbm, f)
# CatBoost
with open(os.path.join(MODEL_DIR, 'CatBoost_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(model_cb, f)
# Stacking MRL
with open(os.path.join(MODEL_DIR, 'StackMLR_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(stacker_mlr, f)
# Stacking XGBoost
with open(os.path.join(MODEL_DIR, 'StackXGB_mfp2_1024.pkl'), mode='wb') as f:
pkl.dump(stacker_xgb, f)最後に
今回は、Morgan Fingerprintと勾配ブースティング系アルゴリズムでモデルを構築する方法を紹介しました。
Morgan FingerprintではなくRDkit記述子を使えばもう少し精度は出ると思います。興味のある方はぜひ試してみてください。
次回のモデル構築編2回目は、グラフニューラルネットワーク系モデルの構築を試したいと思います。




コメント