事前学習モデルとして「CheMeleon」を利用し、物性予測モデルの構築/評価を行います。
実行環境
- OS: Ubuntu 22.04.3 LTS
- CPU: AMD Ryzen 7 5700X 8-Core Processor
- GPU: NVIDIA GeForce RTX 3060 Ti (8G)
- RAM: 32GB
CheMeleon について
CheMelonは、2025年に発表された「分子記述子を利用した教師あり事前学習モデル (descriptor-based pretraining model) 」です。
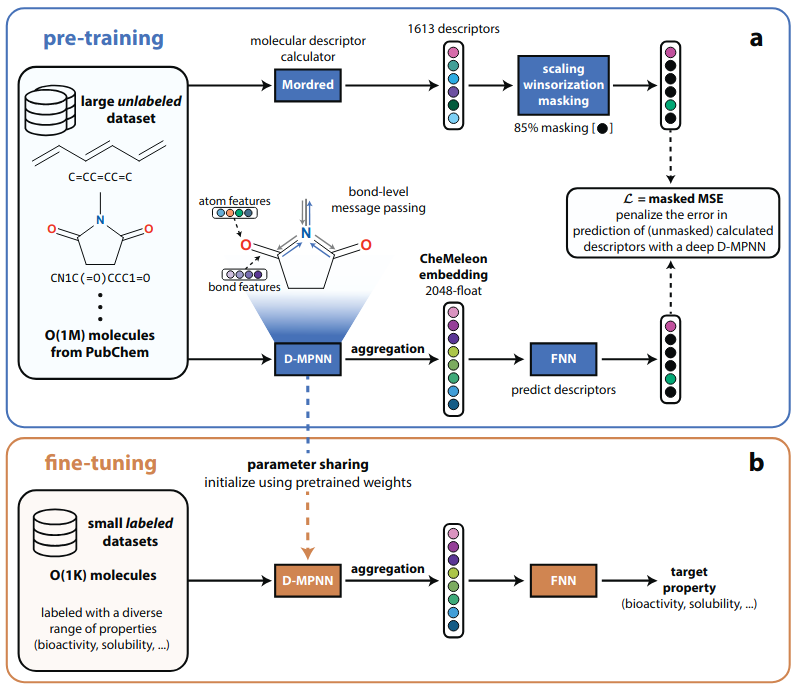
教師ありの事前学習ではラベル付きのデータセットが必要となりますが、高品質なデータの用意には非常にコストがかかります。
分子表現においても実験値や量子化学計算値を利用した事前学習モデルが主流ですが、ラベル付きデータの施設間誤差や、教師なしの事前学習モデルと比べて考慮できる化学空間が狭い、といった課題があります。
CheMeleonでは、ノイズが少ない分子記述子(Mordred記述子)を利用することで表現学習の安定化を図っており、他の事前学習モデルと比較して少量のデータ数(約100万)で下流タスクの高い予測精度を達成しています。
今回は、物性予測に対してCheMeleonの分子表現がどの程度有用かを確認してみます。
環境構築
CheMeleonは、Directed message passing neural network (D-MPNN) を利用して分子表現を学習しています。
D-MPNNは「Chemprop」というオープンソースソフトウェアで利用可能であり、CheMeleonの学習済みモデルにも対応しています。
そのため、今回はChempropからCheMeleonを利用したモデル構築を行いたいと思います。
以下、私の環境で実際に利用したコマンドです。モデル構築のためにjupyterもインストールします。
cd (任意のディレクトリ)
git clone https://github.com/chemprop/chemprop.git
cd chemprop
conda env create -f environment.yml
conda activate chemprop
pip install -e .
conda install jupyter次に、CheMeleonの学習済みモデルをダウンロードします。こちらのサイトからダウンロードして、任意のフォルダに設置しましょう。
以上で環境構築は完了です。
物性予測モデルの構築/評価
Chempropのリポジトリ上にファインチューニング用のnotebookが公開されています。
こちらを参考にして、ニューラルネットワークのアーキテクチャが同じ3種のモデルを比較します。
- デフォルト設定(ゼロベースの学習)
- ファインチューニングA(CheMeleonの学習済みパラメータを固定;CheMeleonの論文と同じ設定)
- ファインチューニングB(CheMeleonの学習済みパラメータを固定しない)
MolecularNetの「Lipophilicity(LogD)」を訓練/検証/テストデータが「8:1:1」になるように分割し、モデルを構築/評価しました(コード)。結果は以下の通りです。
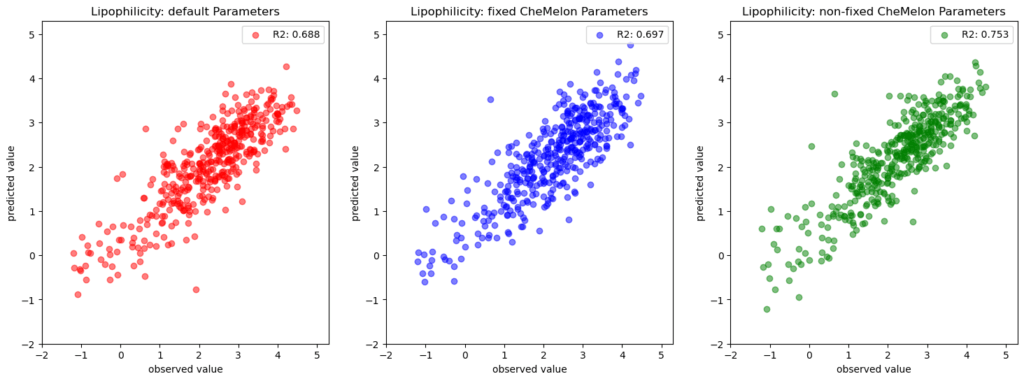
左から、1. デフォルト設定、2. ファインチューニングA、3. ファインチューニングBです。
1.よりも2.の精度が大きく上がると考えていましたが、予想と違いそんなに精度の違いが認められませんでした。LogDが予測しやすくて差が出なかったんでしょうか?
3.が一番精度が出ており、”ある程度化学的性質に合わせた重み付けからスタートした方が学習が早い”、ということを示しているのだと思われます。ただし、過学習感は否めないのでより丁寧な検証が必要そうです。
今回のちょっとした検証からは有用性があまりわかりませんでしたが、CheMeleonは汎化性能が高い分子表現として期待できます。色々と試してみる価値はありそうです。
最後に
CheMeleonを利用して物性予測モデルの構築と評価を行いました。
今回の検証を通して、Chempropの使い勝手が非常に良いことを実感しました。他にの色々と機能があるので、上手に使いこなして行きたいと思います。




コメント