創薬向けOSS「Matcher」をローカル環境に実装します。
実行環境
- OS:WSL2, Ubuntu24.04 (Windows 11 Pro)
- CPU: Intel(R) Core(TM) Ultra 9 185H (WSL2プロセッサ数; 22コア)
- GPU: NVIDIA GeForce RTX 4070 (8GB)
- RAM: 32GB
- Docker: 28.5.0
Matcher について
Matcher は、Merck社が公開しているオープンソースの MMPA(Matched Molecular Pair Analysis)用 Web アプリケーションです。
バックエンドにはRDKitベースのmmpdbを利用し、化合物構造とプロパティから置換ルールや差分統計を生成します。
ユーザーはブラウザ上で直感的にMMPを検索・可視化でき、SAR解析やリード最適化の判断を支援します。
Dockerを用いたローカル実装に対応しており、研究室や企業内で独自データを安全に活用できるのが大きな利点です。
環境構築
Githubの記載に従って、Dockerを用いた環境構築を行います(Dockerの導入方法は割愛します)。
まずはリポジトリをローカル環境にクローンしましょう。
cd (任意のディレクトリ)
git clone https://github.com/Merck/matcher.git次に、バックエンドに利用されているサブモジュールを反映させます。リポジトリのディレクトリへ移動して、以下のコマンドを実行しましょう。
cd matcher
git submodule update --init --recursiveこれで、backend/mmpdb の中身(matcher-mmpdb)がローカル環境にもコピーされました。
私の環境(2025年10月現在)では、フロントエンドの環境においてnumpyとpandasのバージョン競合が確認されました。
こちらを解消するために、frontend/requirements.txt にnumpyのバージョンを指定する記述を追加しました。
echo "numpy==1.23.5" >> frontend/requirements.txt最後に、以下のコマンドでコンテナを起動します。
docker compose build --no-cache
docker compose up上手くいった場合、ブラウザ上で「http://localhost:8000」を入力するとWebアプリが起動します。
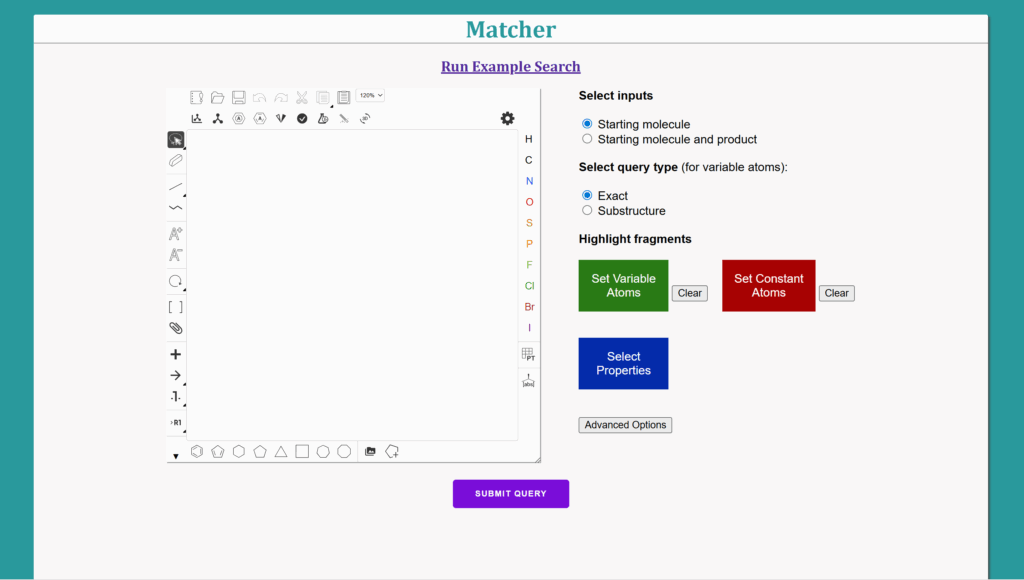
コマンドライン上でdocker composeが起動している状態であれば、Webアプリをいつでも利用可能です。
バックグラウンドで起動したい場合は、control+C でジョブを終了した後に以下のコマンドを実行しましょう。
docker compose stop
docker compose up -d最後に
創薬向けのOSS「Matcher」をローカル環境に実装しました。
今回のようなデフォルトの起動では、クイックスタート用のデータ(ChEMBL由来の1089個)が検索対象になります。
任意のデータを利用するためには、mmpdbを利用してデータベースを作成する必要があるのでご注意ください。




コメント