創薬向けのOSSである「ED2Mol」をローカル環境で動かしてみたいと思います。
実行環境
- OS:WSL2, Ubuntu24.04 (Windows 11 Pro)
- CPU: Intel(R) Core(TM) Ultra 9 185H (WSL2プロセッサ数; 22コア)
- GPU: NVIDIA GeForce RTX 4070 (8GB)
- RAM: 32GB
ED2Mol について
ED2Molは、2024年12月に公開された分子生成モデルです。
X線結晶構造解析やクライオ電子顕微鏡によって得られる電子密度マップを活用する点が大きな特徴であり、生成された分子の物理化学的妥当性において、TargetDiff や Pocket2Mol などの既存モデルと比較して優れた性能を示しています。
今回はこのED2Molについて、実行環境の構築及びサンプルデータを利用したテストを行いたいと思います。
環境構築
Githubの記載に従って、mambaを利用してconda環境を作成します。
mambaは、condaと互換性のあるパッケージマネージャで、RustとC++で実装されているため、condaよりも高速に動作します。
mamba自体はcondaコマンドで導入でき、通常はbase環境にインストールして利用します。
conda install mamba -n base -c conda-forge公式のリポジトリには、①マニュアルのインストール方法と②yamlファイルからのインストール方法が記載されていますが、今回は②の方法でインストールを行います。
まずは、ローカル環境にリポジトリのクローンを作成しましょう。
cd (任意のディレクトリ)
git clone https://github.com/pineappleK/ED2Mol.git次に、mambaを利用して「ed2mol」という名前のconda環境を作成します。リポジトリにyamlファイルがアップロードされているため、クローンしたディレクトリに移動しコマンドを実行しましょう。
cd ED2Mol
mamba env create -f ed2mol_env.yml -n ed2mol
mamba activate ed2molED2Molでは訓練済みモデルが利用可能です。以下のコマンドでファイルをダウンロードしましょう。
wget https://github.com/pineappleK/ED2Mol/releases/download/v1.0/weights.zip
unzip weights.zip以上で環境構築は完了です。
ED2Mol の操作
ED2Molでは、①De novo生成と②ヒット最適化、2つの用途で分子生成が可能です。
リポジトリ内のconfigsフォルダ内には、サンプルデータの分子生成に利用可能な設定ファイル(.yaml)があります。今回はこちらを利用してDe novo生成を実施してみたいと思います(ただし、output_mols_num は 10 に変更)。
Githubの記載通り、以下のコマンドを実行します。
CUDA_VISIBLE_DEVICES="0" python Generate.py --config ./configs/denovo.yml私の環境では2時間弱かかりましたが、10個の分子が生成されました(途中でエラーメッセージが出て止まりましたが、結果がそれっぽかったので今回は気にしないこととします)。
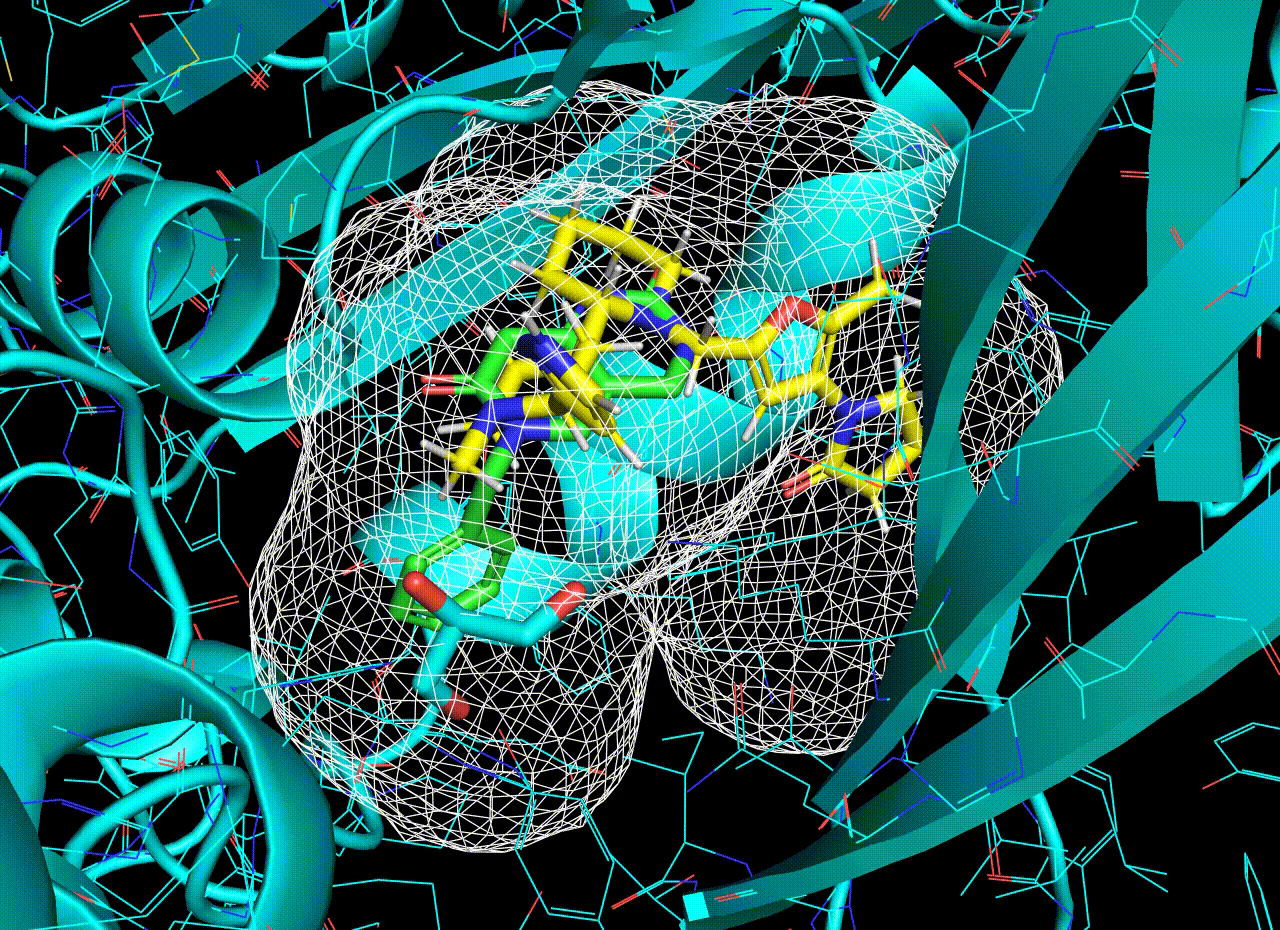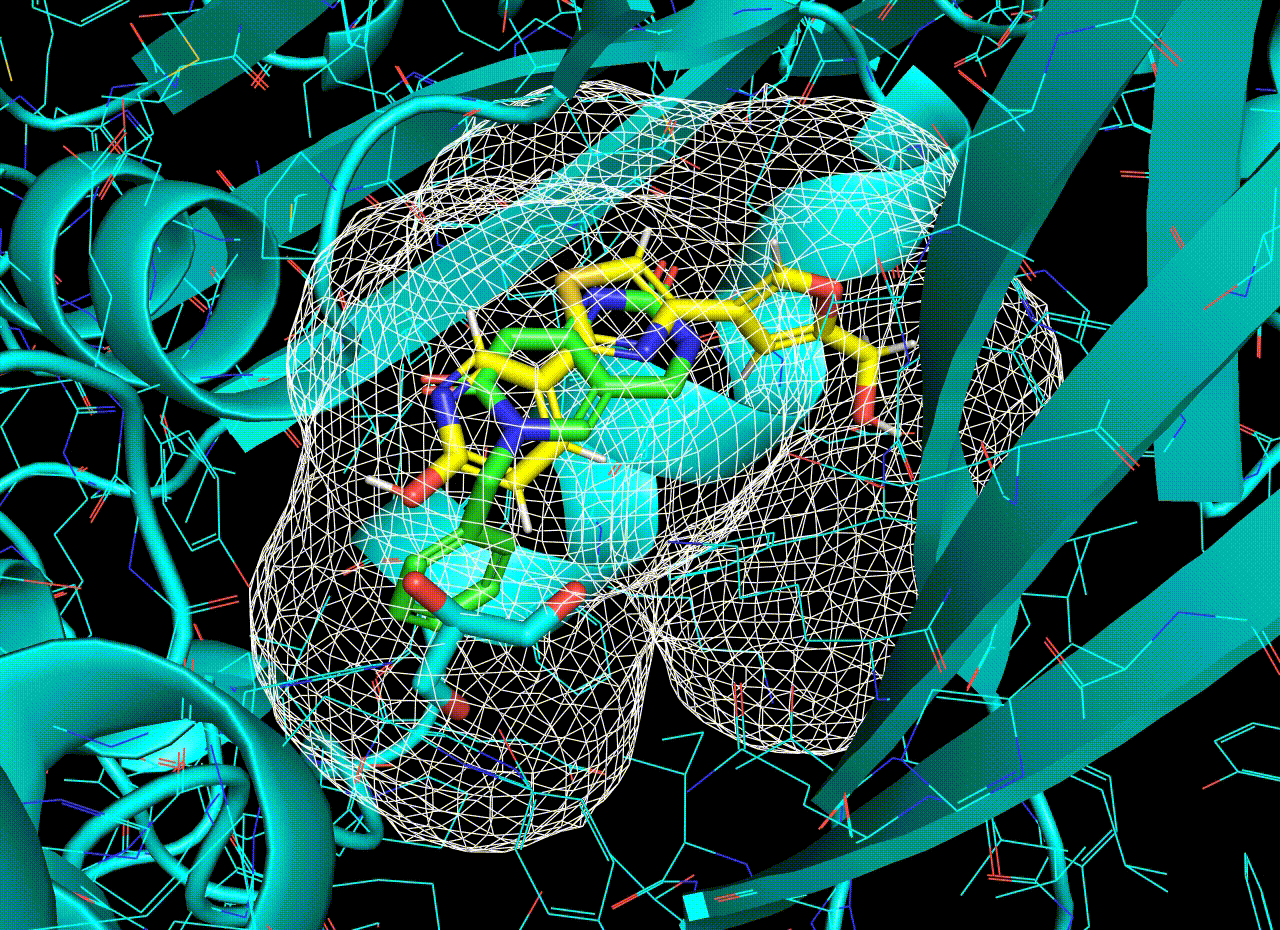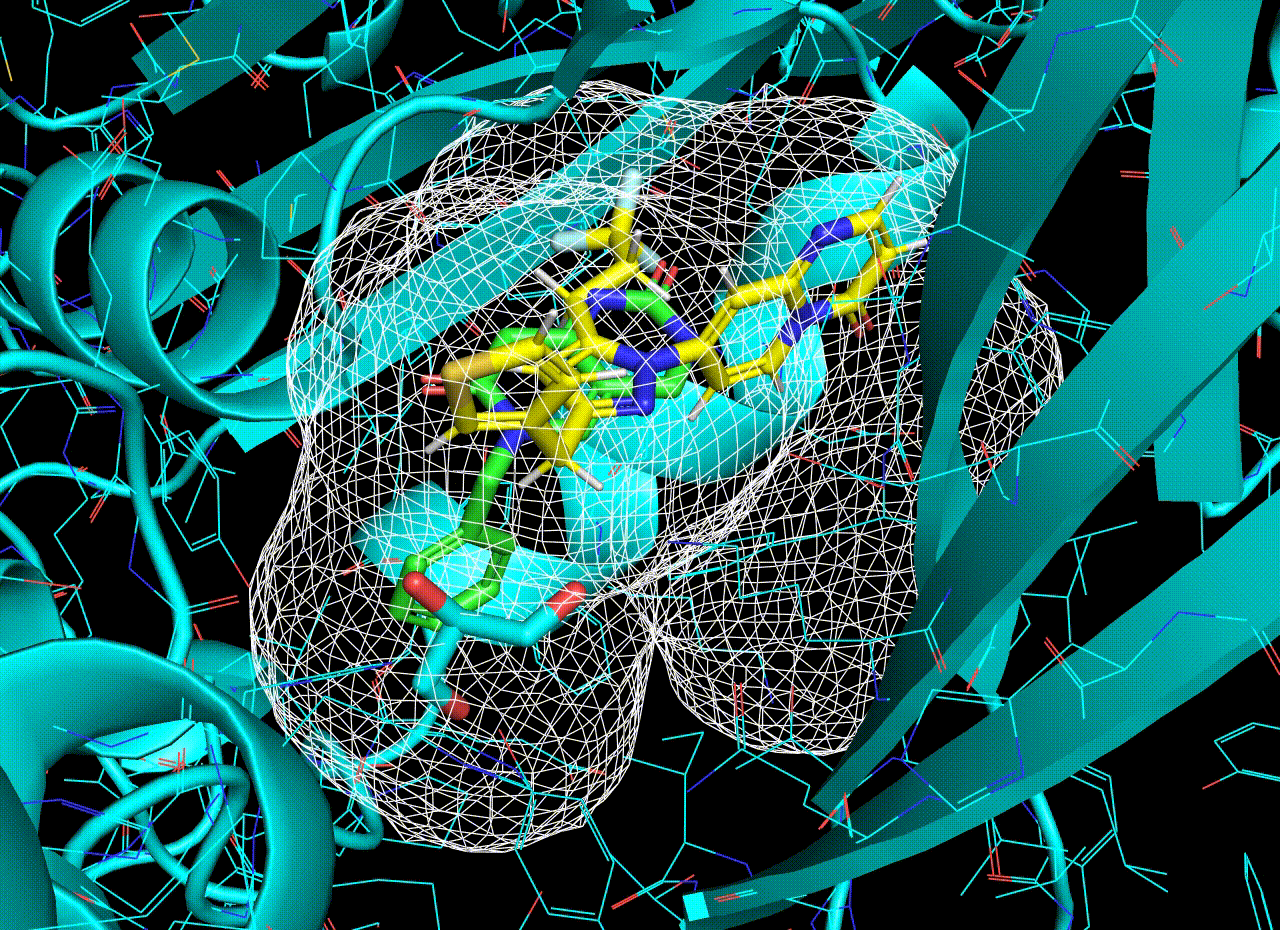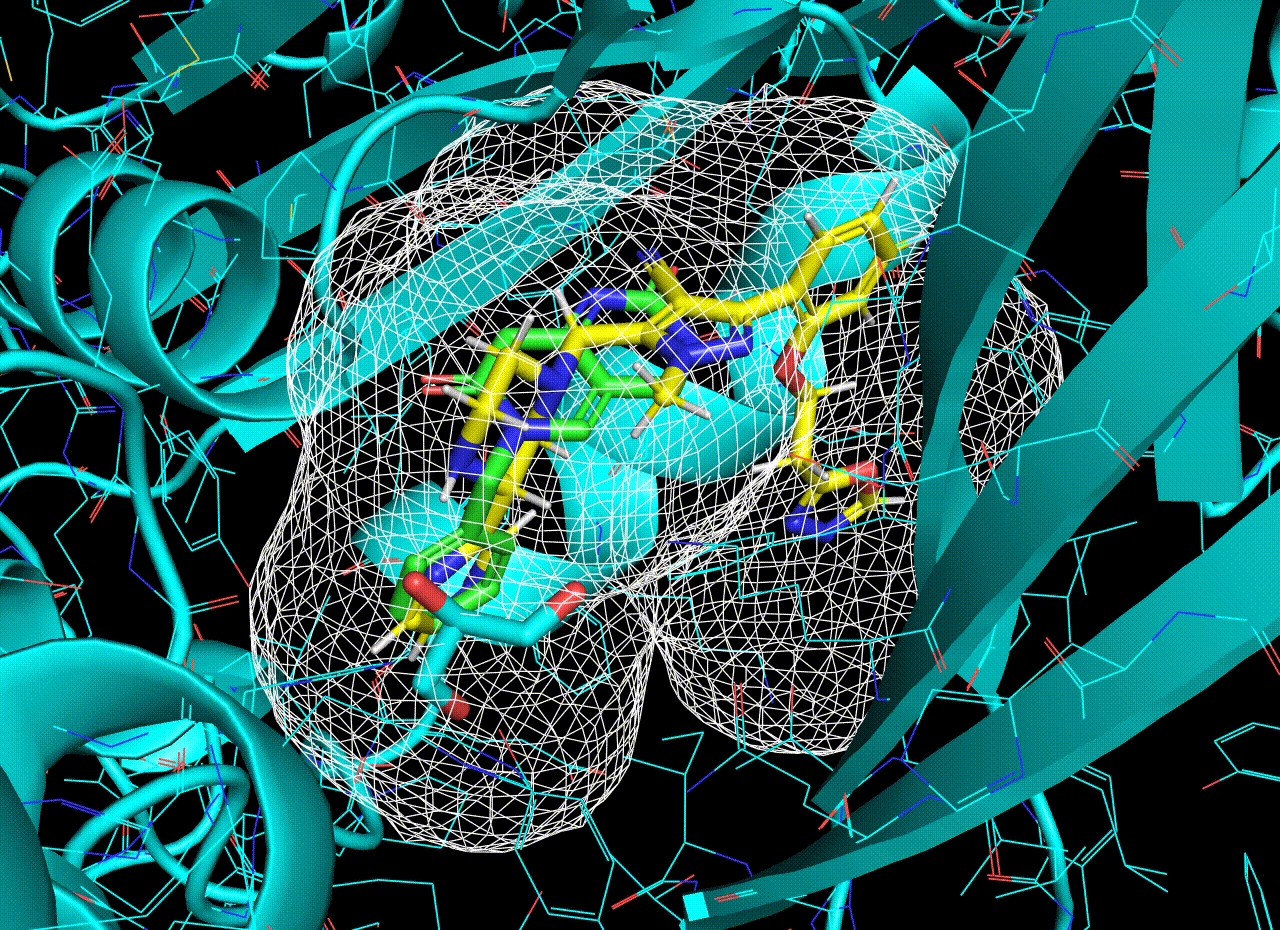
結果について、PyMOLで可視化してみたいと思います。緑色がサンプルデータのリガンド、黄色がDe novo生成されたリガンドです。また、白メッシュはリガンドの電子密度(予測値)です。
リガンドの電子密度は結合ポケットの電子密度から推定されたものです。分子生成はこの予測電子密度の内側に収まるよう行われています。
最後に
創薬向けのOSSである「ED2Mol」をローカル環境で動かしてみました。
合成容易性や結合親和性については(分からないので)割愛しますが、リガンドのコンフォメーションについてはかなり良い印象です。
私は3D分子の生成モデルは DiifLinker と DiffDec くらいしか触ったことがありませんが、それらと比べると安定なコンフォメーションのように感じます。
強化学習系の2D分子生成モデルと比べてどのような違いが見られるか、確認してみたいですね。




コメント