前回の続きです。クレンジングしたデータを使って予測モデルを構築していきます。
予測モデル構築編の2回目は、グラフニューラルネットワーク系アーキテクチャの1つである「Attentive FP」を用いてモデルを構築します。
Graph Neural Network(GNN)について
グラフニューラルネットワーク(Graph Neural Network:GNN)は、グラフ構造のデータを取り扱うニューラルネットワークです。
グラフ構造について
「グラフ(Graph)」とは、データの要素(ノード)と、それらをつなぐ関係(エッジ)から成るデータを指します。従来型の機械学習が取り扱いを得意とする「表形式データ(Tabular data)」とは異なるデータの表現方法です。
化合物は、原子をノード、結合をエッジとみなしたグラフ構造のデータ(分子グラフ)として表現することができます。
GNN について
GNNでは、ノード間の情報を伝達・更新する「メッセージパッシング」と、グラフ全体の特徴を集約する「リードアウト」を通じて、ノードやグラフの表現を学習します。
教師あり学習では、タスクに応じてノードの特徴を周囲の関係性とともに学習し、ノード単位またはグラフ全体の特徴を適切に捉えることが期待されます。教師なし学習では、ラベルなしのデータからノードやグラフの構造を捉え、有用な特徴表現を学習することが期待されます。
従来の分子の表現(Morgan Fingerprintなど)は人間が設計したルールベースの手法であり、情報の欠落や汎用性の問題がありましたが、GNNを使うことでより柔軟で精度の高い特徴量を得ることができるのでは?と期待されています。
Attentive FPについて
Attentive FPは、分子グラフを扱うための Graph Neural Network(GNN) の一種です。
Attentive FPは、注意機構(Attention)によってノード間の重要度を学習し、重要なノードの情報に重みを付けることが特徴です。
また、メッセージパッシングとリードアウトにGRU(Gated Recurrent Unit)を組み込むことで、”近接する原子”と”遠方の原子”の双方を考慮した上で重要な関係性を保持する設計となっています。
2020年に発表されたアーキテクチャですが、強力ながらそこまで計算量が多くなく、かつハイパーパラメータをいじらなくてもそこそこ精度がでるため、管理人は現在も重宝して使っています。
今回はこのAttentive FPを「DGL-LifeSci」というライブラリを使って実装していきたいと思います。
モデル構築の流れ
「DGL-LifeSci」はライフサイエンス分野に特化したGNN用ライブラリです。
「DGL(Deep Graph Library)」というGNN向けフレームワークをベースとしており、化合物の物性予測をサポートする関数が充実しています(AWSのチームが開発していますが、最終更新が何年も前なので今後アップデートはないかもしれません)。
GNNモデルを構築するのであれば、「DeepChem」や「Chemprop」の方が機能もサンプルも充実していて覚える価値があるのかもしれません。ですが、初学者レベルには環境構築が難しく、モデル構築のイメージがつきにくいように感じました。
そこで今回は、GNNによるモデル構築の基礎を身につけられるように、”比較的簡単にGNNが実装できる、一方でGNNの仕組みを理解していないとモデル構築が難しい”、DGL-LifeSciを利用したいと思います。
モデル構築は以下の流れで行います。
- データセットの準備
- ネットワークの構築
- モデルの訓練と評価
データセットの準備
GNNを利用するためにはインプットである化合物の構造情報をDGLに対応したグラフ(DGLGraphオブジェクト)に変換する必要があります。
化合物のデータ構造はRDkitを使えば大抵どうにかなりますが、インプット用のグラフ構造へ変換するためには複雑な処理が必要です。
また、DGL-LifeSciはPytorchをベースとしてディープラーニングモデルを構築しています。そのため、データをPytorchが扱いやすい形式(Datasetオブジェクト)に変換してやる必要があります。
DGL-LifeSciでは、構造情報のDGLGraphオブジェクト化からPytorchのDatasetオブジェクトへの変換までを簡単に実装することができるので、まずはこちらを実行してDatasetオブジェクトを作成します。
次に、作成したDatasetオブジェクトをDataLoaderオブジェクトに設定します。
ディープラーニングでは、正則化や計算資源節約などの観点から「ミニバッチ学習」という手法を用いるのが一般的です。PytorchのDataLoaderを使うことでデータのバッチ処理が簡単になるため活用していきます。
DGL-LifeSciでは、DataLoaderオブジェクトの設定を行う機能はない(はずの)ため、こちらはPytorchのモデル構築の方法に従い実装していきたいと思います。
ネットワークの構築
DGL-LifeSciではメッセージパッシングとリードアウトの実装が充実していますが、モデルを構築するためにはそれらを組み合わせたネットワークを構築する必要があります。
また、リードアウトで獲得したグラフの特徴量をタスクと結びつけるネットワーク(アウトプット層)は自分で構築する必要があります。
今回は、アウトプット層を自分で定義すると共に、DGL-LifeSciが提供するメッセージパッシング層、リードアウト層とつなぎ合わせたモデルを構築したいと思います。
モデルの訓練と評価
ディープラーニングモデルの訓練は、誤差逆伝播法(バックプロパゲーション)によるパラメータ最適化を繰り返すことで行います。
この最適化は非常に重要な過程であり、適切に管理しなければモデルが過学習してしまい未知データに対する予測が難しくなってしまいます。
Pytorchのモデルは訓練過程を自分で実装する必要があります。表現方法は多岐に渡りますが、今回は”Early Stopping”という過学習を回避する実装を組み込んで訓練していきたいと思います。
実装
環境構築
ライブラリの依存性はDGL-LifesciのGithubに記載されていますが、選択肢が幅広いので管理人が構築したときの依存性とパッケージ管理の方法を以下に記載します。
- python 3.8
- torch 2.1.0+cu118 (pip)
- torchaudio 2.1.0+cu118 (pip)
- torchvision 2.1.0+cu118 (pip)
- DGL 2.4.0+cu118 (pip)
- DGL-LifeSci 0.3.2 (pip)
- RDkit 2024.3.5 (pip: default) ※JTVAEは使わないので推奨バージョンを無視
- jupyter 1.1.1 (conda: default) ※Jupyter Notebookを利用するため
- matplotlib 3.7.3 (conda: default) ※プロットを作成するため
OSがUbuntuなら下記のコマンドで再現できるのではないでしょうか?
conda create -n dgllife python=3.8
conda activate
pip install torch==2.1.0 torchvision==0.16.0 torchaudio==2.1.0 --index-url https://download.pytorch.org/whl/cu118
pip install dgl -f https://data.dgl.ai/wheels/torch-2.1/cu118/repo.html
pip install dgllife
pip install rdkitGPUの利用を想定していますが、GPUない場合でも上記の環境構築で問題ない(はず)です。
PytorchはCUDA Toolkit及びcuDNNを含むインストー方法なので、NVIDIAドライバーさえ設定していればGPUを認識してくれると思います。
ライブラリがインストールできたら、以下のコードを実行してGPUが利用可能な確認してみましょう。GPUが利用可能であれば”True”が表示されます。
import torch
print(torch.cuda.is_available())分子グラフの作成
SMILESからDGLGraphオブジェクトを生成する設定
まずは、化合物の構造からDGLGraphオブジェクトを作成するための設定を行います。今回はDGL-Lifesciが提供する以下のクラスを利用します。
- dgllife.utils.mol_to_grap.SMILESToBigraph
- dgllife.utils.featurizers.AttentiveFPAtomFeaturizer
- dgllife.utils.featurizers.AttentiveFPBondFeaturizer
1番目は「SMILESをDGLGraphに変換するクラス」です。インスタンス化の際にノード(原子)とエッジ(結合)の特徴量計算方法を指定することができます。
2番目と3番目はそれぞれ「AttentiveFP用のノードの特徴量を計算するクラス」と「AttentiveFP用のエッジの特徴量を計算するクラス」です。これらをインスタンス化し1番目のクラスをインスタンス化する際に指定することで、SMILESから学習用のDGLGraphを生成することが可能になります。
実際に利用するコードは以下の通りです。これにより”smi_to_g”というクラスインスタンスにSMILESを与えることでDGLGraphが返ってくるようになります。
from dgllife.utils.featurizers import AttentiveFPAtomFeaturizer, AttentiveFPBondFeaturizer
from dgllife.utils.mol_to_graph import SMILESToBigraph
# ノード(原子)とエッジ(結合)に特徴量を与えるクラスをインスタンス化
atom_featurizer = AttentiveFPAtomFeaturizer(atom_data_field='h') # DGLGraphで"h"という名前で特徴量が登録されるようになる
bond_featurizer = AttentiveFPBondFeaturizer(bond_data_field='e') # DGLGraphで"e"という名前で特徴量が登録されるようになる
# SMILESから分子グラフ(DGLGraph)を作成するクラスをインスタンス化
smi_to_g = SMILESToBigraph(
node_featurizer=atom_featurizer, # ノードに与える特徴量を計算する関数: rdkit.Chem.rdchem.Mol -> dict
edge_featurizer=bond_featurizer, # エッジに与える特徴量を計算する関数: rdkit.Chem.rdchem.Mol -> dict
canonical_atom_order=True, # 原子の並び順をRDkitのcanonicalな表現にするかどうか(デフォルト:True)
explicit_hydrogens=False, # 水素原子を明示的に表現するかどうか(デフォルト:False)
num_virtual_nodes=0, # バーチャルノードの数(デフォルト:0)
add_self_loop=False, # self-loopの設定(デフォルト:False)
)Pytorchモデル用のDatasetを作成
DGL-Lifesciが提供する以下のクラスを利用します。
- dgllife.data.csv_dataset.MoleculeCSVDataset
pandasのDataFrameオブジェクトと先述の”smi_to_g”を指定してインスタンス化することで、PytorchのDatasetオブジェクトが生成します。
import os
import pandas as pd
from dgllife.data.csv_dataset import MoleculeCSVDataset
# データの読み込み
df_train = pd.read_csv('~/shaeo-blog/pj-logd/dataset/train.csv')
df_test = pd.read_csv('~/shaeo-blog/pj-logd/dataset/test.csv')
# キャッシュ先のパスを設定する
HOME = os.getenv('HOME')
train_cache_path = HOME + '/shaeo-blog/pj-logd/dataset/dglgraph_train.bin'
test_cache_path = HOME + '/shaeo-blog/pj-logd/dataset/dglgraph_test.bin'
# 分子グラフと目的変数を含むDatasetを作成
dataset_train = MoleculeCSVDataset(
df=df_train, # SMILESと目的変数のデータフレーム
smiles_to_graph=smi_to_g, # SMILESからDGLGraphを生成する関数
smiles_column='canonical_smiles', # SMILESの列名
cache_file_path=train_cache_path, # キャッシュ先のファイルパス
task_names='standard_value', # 目的変数の列名
load=False, # キャッシュを利用するかどうか(デフォルト:False)
init_mask=False, # データの有無を表すマスクデータを作成するかどうか。マルチタスクで有用
n_jobs=-1, # 利用するCPUのコア数(-1で全CPUを利用する)
)
dataset_test = MoleculeCSVDataset(
df=df_test,
smiles_to_graph=smi_to_g,
smiles_column='canonical_smiles',
cache_file_path=test_cache_path,
task_names='standard_value',
load=False,
init_mask=False,
n_jobs=-1,
)このDatasetオブジェクトには、SMILES, DGLGraph, standard_valueを要素とするタプルが格納されており、インデックスを指定することで対応したデータが確認できます。
以上でDatasetオブジェクトが用意できましたが、今回は訓練時にEarly Stoppingを設定するため検証用データを作成しておきましょう。
# 検証用データを作成する(EarlyStopping用)
from torch.utils.data import Subset
# シャッフルしたインデックスを取得
indices = torch.randperm(len(dataset_train)).tolist()
# データセットを分割
train_size = int(0.8 * len(dataset_train))
val_size = int(0.2 * len(dataset_train))
train_indices = indices[:train_size]
val_indices = indices[train_size:]
dataset_train_a = Subset(dataset_train, train_indices)
dataset_train_b = Subset(dataset_train, val_indices)DataLoaderオブジェクトの作成
作成したDatasetをDataLoderに読み込ませます。
DataLoaderはイテラブルなオブジェクトであり、設定したバッチサイズ単位でバッチ化されたデータを取り出すことができます。
ただし、DataLoaderのデフォルト設定ではDGLGraphのバッチ処理に対応できないため、バッチ処理の対応させるための関数を定義する必要があります。
以下のコードで、DGLGraphのバッチ処理に対応したデータ関数を定義し、DataLoaderオブジェクトを作成しましょう。
# PytrochのDataLoaderクラスを作成
import dgl
from torch.utils.data import DataLoader
# DataLoaderから取り出すデータを設定する関数
def collate(data):
_, g, y = zip(*data) # SMILES, DGLGraph, 目的変数ごとにまとめ直す
bg = dgl.batch(g) # DGLGraphのバッチ化
y = torch.stack(y).view(-1, 1) # 0次元型のテンソルを要素に持つリスト→2次元のテンソル(N, 1)
return bg, y
# DataLoaderの作成
dataloader_train = DataLoader(
dataset=dataset_train_a, # PytorchのDatasetインスタンス
batch_size=64, # 1バッチあたりのサンプル数
shuffle=True, # サンプルの順序を入れ替えるかどうか(デフォルト:False)
collate_fn=collate, # サンプルの取り出し方を定義
pin_memory=False, # 計算の高速化に関わる処理?(デフォルト:False)
num_workers=0, # 使用するCPUコア数(デフォルト:0)
)
dataloader_valid = DataLoader(
dataset=dataset_train_b,
batch_size=64,
collate_fn=collate,
pin_memory=False,
num_workers=0,
)
dataloader_test = DataLoader(
dataset=dataset_test,
batch_size=64,
shuffle=False,
collate_fn=collate,
pin_memory=True,
num_workers=0,
)ネットワークの構築
ネットワークの設定
メッセージパッシング層とリードアウト層は、DGL-LifeSciが提供している以下のクラスを利用します。
- dgllife.model.gnn.attentivefp.AttentiveFPGNN
- dgllife.model.readout.attentivefp_readout.AttentiveFPReadout
2クラスともインスタンス化の際に、インプットの次元数やネットワークの条件(総数やドロップアウトの割合など)を指定することで、タスクに適したネットワークを生成することができます。
インスタンス化で指定できる引数に関しては、コードのコメントアウトに記述したのでご参考ください。
また、リードアウト以降のネットワークは自分で定義する必要があります。
Pytorchのネットワークは”torch.nn.Module”クラスを継承することで簡単に作成することができます。この辺りの話は難しくなるので詳細は書きませんが、コードのような記述をすることで任意のネットワークが生成できると考えてください。
以下、ネットワークを設定するコードです。
# ネットワークの設定
import torch.nn as nn
from dgllife.model.gnn.attentivefp import AttentiveFPGNN
from dgllife.model.readout.attentivefp_readout import AttentiveFPReadout
# 出力層を定義する
class OutputNN(nn.Module):
# 設定
def __init__(
self,
input_feat_size, # リードアウト後の特徴量次元数
dropout=0.2 # ドロップアウトの割合
):
super(OutputNN, self).__init__()
self.output = nn.Sequential(
nn.Linear(input_feat_size, input_feat_size), # リードアウト後の特徴を線形変換
nn.ReLU(), # ReLUで非線形変換
nn.Dropout(dropout), # ドロップアウト
nn.Linear(input_feat_size, 1) # 1次元に線形変換(予測値)
)
def forward(self, graph_feat):
output = self.output(graph_feat) # 上記で定義したネットワークを利用し予測値を得る
return output # 予測値を返す
# AttentiveFPを定義する
class AttentiveFP(nn.Module):
# 設定
def __init__(
self,
gnn_node_feat_size, # ノードの特徴量の次元数
gnn_edge_feat_size, # エッジの特徴量の次元数
gnn_num_layers=2, # メッセージパッシングの回数
gnn_dropout=0., # メッセージパッシング層でのドロップアウトの割合
graph_feat_size=200, # リードアウト後のグラフの特徴量の次元数
readout_num_timesteps=2, # リードアウトに利用するGRUの層数
readout_dropout=0., # リードアウト層でのドロップアウトの割合
output_dropout=0.2 # アウトプット層でのドロップアウトの割合
):
super(AttentiveFP, self).__init__()
# メッセージパッシング層
self.gnn = AttentiveFPGNN(
node_feat_size=gnn_node_feat_size,
edge_feat_size=gnn_edge_feat_size,
num_layers=gnn_num_layers,
graph_feat_size=graph_feat_size,
dropout=gnn_dropout
)
# リードアウト層
self.readout = AttentiveFPReadout(
feat_size=graph_feat_size,
num_timesteps=readout_num_timesteps,
dropout=readout_dropout,
)
# アウトプット層
self.output = OutputNN(
input_feat_size=graph_feat_size,
dropout=output_dropout
)
# ニューラルネットワークの流れ
def forward(self, g, node_feats, edge_feats):
node_feats = self.gnn(g, node_feats, edge_feats) # メッセージパッシング
g_feats = self.readout(g, node_feats, get_node_weight=False) # リードアウト
output = self.output(g_feats) # アウトプット
return output # 予測値を返すOutputNNクラスは自分で定義したアウトプット層のネットワークで、AttentiveFPクラスは、AttentiveFPGNNクラス、AttentiveFPReadoutクラス、OutputNNクラスをつなげた最終的なネットワークになります。
Pytorchのモデルは、forward関数で順伝播の計算が走ります。forward関数の処理は、クラス内でインスタンス化した各層に対してグラフデータを渡し、1次元データのアウトプット(予測値)を算出するものとなっています。
ネットワークのインスタンス化と各種設定
設定したネットワークをインスタンス化すると共に、最適化手法やEarly Stoppingの設定を行います。
まず、ネットワークのインスタンス化は以下のコードで行います。
絶対に必要となる引数は”gnn_node_feat_size”と”gnn_edge_feat_size”です。それぞれ先述したクラスから値を求められるのでそちらを利用します。
# ネットワークのインスタンス化
model = AttentiveFP(
gnn_node_feat_size=atom_featurizer.feat_size(), # ノードの特徴量次元数を指定
gnn_edge_feat_size=bond_featurizer.feat_size() # エッジの特徴量次元数を指定
)今回のモデルでは、損失関数としてMSE (Mean Squared Error:平均二乗誤差)、最適化アルゴリズムとしてAdamを使用します。
実装は以下のコードです。どちらもPytorchで用意されているクラスを利用します。学習率がハイパーパラメータですが今回は”0.001”とします。
# 最適化手法の定義
import torch.optim as optim
criterion = nn.MSELoss() # MSEを最小化する
optimizer = optim.Adam(model.parameters(), lr=0.001) # lr は 学習率また、今回のモデルでは過学習を防ぐために「Early Stopping」という手法を使用します。
ディープラーニングでは過学習を防ぐために、訓練データとは別途検証データを用意して”検証データの精度に改善が認められなくなった段階で学習をやめる”といったことをします。
Early Stoppingは、Pytorch単体では自力で実装する必要がある一方、DGL-LifeSciでは以下のクラスとして提供されています。
- dgllife.utils.early_stop.EarlyStopping
今回はこちらを用いて「検証データに20エポック改善が認められなければ学習を中断する」といった処理を追加したいと思います。
実装は以下の通りです。検証データで最も精度が良かったエポックのモデルを保存するために、パスを指定する必要があります。
# EarlyStoppingの定義
from dgllife.utils.early_stop import EarlyStopping
MODEL_DIR = HOME + '/shaeo-blog/pj-logd/model/AttentiveFP' # モデルの保存先のディレクトリ
os.makedirs(MODEL_DIR, exist_ok=True) # モデルの保存先を作成
stopper = EarlyStopping(
mode='lower', # 与えられた値が低い方が良いモデルだと判断する
patience=20, # 与えられた値が最良のものから20回変わらなければフラグを建てる
filename=MODEL_DIR+'/best_model.pth' # ベストモデルの情報を保存するためのパス
)各種設定の最後にデバイスを設定します。GPUを利用した学習では、GPU計算を行いたいパラメータ(変数)を明示的にGPUへ送る必要があります。
以下のコードは、GPUが利用できる場合はGPUを、できない場合はCPUを使う設定にするための処理です。GPUが利用可能な場合はモデルをGPUに移動します。
# デバイスの設定
device = 'cuda' if torch.cuda.is_available() else 'cpu'
print(device)
# モデルをGPUに移動する
if device == 'cuda':
model.to(device)以上でモデルが訓練可能な状態となりました。モデルのネットワーク構造はモデルの変数をprintすることで確認できるので興味のある方は確認してみてください。
モデルの訓練と評価
訓練モードとテストモードの設定
Pytrochのモデルには、訓練モードと推論モードがあります。
これらのモードでは、勾配計算の有無やドロップアウトの挙動に違いがあり、適切に切り替えなければ正しいモデル構築ができません。
今回は、訓練モードと推論モードを共に関数化して利用します。訓練モードの関数はモデルのパラメータ更新にのみ利用し、テストモードの関数は予測値の算出にのみ利用します。
実際に利用する関数を定義したコードは以下のとおりです。
# 訓練とテストの設定
# 訓練モードの定義
def model_train(model, dataloader_train, device):
model.train() # 訓練モードに切り換える
# モデルを訓練する
for (g, y) in dataloader_train:
# GPUに移動する
if device == 'cuda':
g, y = g.to(device), y.to(device)
# 学習
optimizer.zero_grad() # 勾配をゼロにリセット
y_pred = model(g, g.ndata['h'], g.edata['e']) # 順伝播
loss = criterion(y_pred, y) # 損失を計算
loss.backward() # 逆伝播
optimizer.step() # 最適化
# GPUからCPUに移動する
if device == 'cuda':
g, y, y_pred = g.to('cpu'), y.to('cpu'), y_pred.to('cpu')
# 変数の削除
del g, y, y_pred
# テストモードの定義
def mode_test(model, dataloader_test, device):
model.eval() # 推論モードに切り替え
list_y = [] # 実測値保存用のリスト
list_y_pred = [] # 予測値保存用のリスト
# 予測値を算出する
with torch.no_grad(): # 勾配計算を無効にする
for (g, y) in dataloader_test:
# GPUに移動する
if device == 'cuda':
g, y = g.to(device), y.to(device)
# 予測値を算出する
y_pred = model(g, g.ndata['h'], g.edata['e'])
# GPUからCPUに移動する
if device == 'cuda':
g, y, y_pred = g.to('cpu'), y.to('cpu'), y_pred.to('cpu')
# 実測値と予測値をリストに追加する
list_y.append(y)
list_y_pred.append(y_pred)
# 変数の削除
del g
# tensorを集約する
y_all = torch.cat(list_y, dim=0)
y_pred_all = torch.cat(list_y_pred, dim=0)
return y_all, y_pred_all
モデルの訓練と予測
いよいよモデルの訓練に入ります。
今回は、最大200エポック(サイクル)を設定して訓練を始めます。Early Stoppingを設定しているので、場合によってはそれよりも早く訓練が終わります。
また、後ほど学習過程を確認できるように各エポックで評価値(MSEとついでにR2)を算出します。
以下、モデルの訓練と予測を実行するコードです。今回はあまり必要ありませんが、特定の周期でモデルを保存しておくのが一般的なようなので20エポックごとにモデルを保存しています。
# モデルの訓練と予測
from tqdm.notebook import tqdm
import torch.nn.functional as F
from sklearn.metrics import r2_score
# エポックの設定
num_epochs = 200
# 評価値保存用のリストを作成
list_y_train_mse = []
list_y_valid_mse = []
# モデルの訓練
for epoch in tqdm(range(num_epochs)):
# モデルの更新
model_train(model, dataloader_train, device)
# 予測値の算出
y_train, y_pred_train = mode_test(model, dataloader_train, device)
y_valid, y_pred_valid = mode_test(model, dataloader_valid, device)
# 評価値の算出
mse_train = F.mse_loss(y_pred_train, y_train).item()
mse_valid = F.mse_loss(y_pred_valid, y_valid).item()
r2_train = r2_score(y_train.numpy(), y_pred_train.numpy())
r2_valid = r2_score(y_valid.numpy(), y_pred_valid.numpy())
print(f'EPOCH {epoch+1}::', 'MSE train: ', f'{mse_train:.5f}', 'MSE valid: ', f'{mse_valid:.5f}', 'R2 train: ', f'{r2_train:.5f}', 'R2 valid: ', f'{r2_valid:.5f}')
# 評価値を保存する
list_y_train_mse.append(mse_train)
list_y_valid_mse.append(mse_valid)
# EarlyStoppingの評価
early_stop = stopper.step(mse_valid, model)
if early_stop:
break
# 20エポックごとにモデルを保存する
if epoch+1 % 20 == 0:
torch.save(model.state_dict(), os.path.join(MODEL_DIR, f'model_epoch{epoch+1}.pth'))
今回は初期シードを固定していないのでばらつきがあると思いますが、Early Stoppingにより100エポック前後で学習が終わったのではないでしょうか?
学習状況を以下のコードで学習状況を確認してみましょう。
# 学習状況を描写する
import matplotlib.pyplot as plt
epochs = list(range(1, len(list_y_train_mse) + 1))
# 学習曲線の描画
plt.plot(epochs, list_y_train_mse, linestyle='-', color='b', label='Train MSE')
plt.plot(epochs, list_y_valid_mse, linestyle='-', color='r', label='valid MSE')
# ラベルとタイトルの設定
plt.xlabel('Epochs')
plt.ylabel('MSE')
plt.title('Learning Curve (MSE over Epochs)')
plt.legend()
# グラフを表示
plt.show()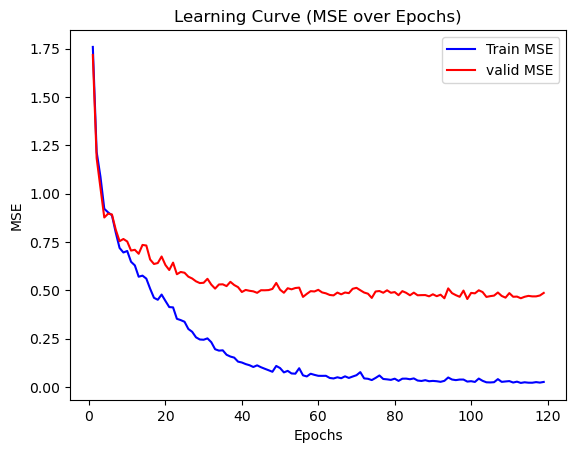
確かにこれ以上学習すると過学習が起こりそうですね。
では最後に学習に使っていないテストデータで評価値(R2)を算出し、モデルがどの程度の精度になったか確認してみましょう。
# テストデータの予測値と評価値を算出する
# ベストモデルのパラメータを読み込む
stopper.load_checkpoint(model)
# テストデータの予測値を算出する
y_test, y_pred_test = mode_test(model, dataloader_test, device)
r2_test = r2_score(y_test.numpy(), y_pred_test.numpy())
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_title('Observed vs Predicted')
ax.set_ylabel('Predicted values of LogD')
ax.set_xlabel('Observed values of LogD')
ax.scatter(y_test, y_pred_test, label=f'AttentiveFP: R2:{r2_test:.3f}', alpha=0.5)
ax.legend()
plt.show()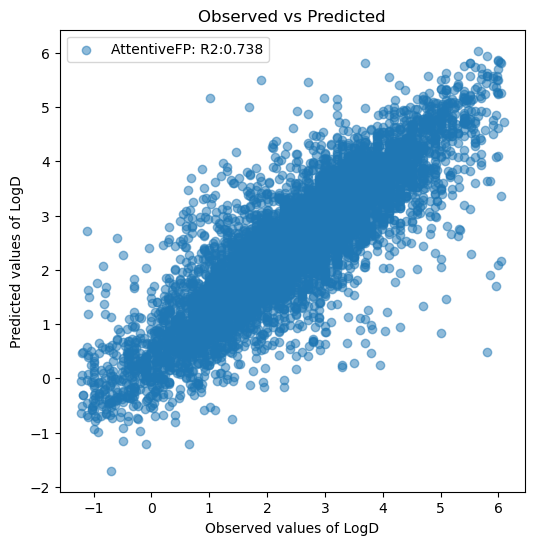
予測精度としてはまだまだですが、前回一番良かったモデルでR2が0.634だったので比べるとかなり精度が良いですね。
面倒ですがハイパーパラメータを調整すればもう少し改善しそうです。予測が大きく外れているサンプルがあるのでAttentive FP単体ではR2>0.8の壁は超えられないと思いますが…。
最後に
今回は、Attentive FPを用いてモデルを構築する方法を紹介しました。
全てのタスクにGNNが適しているわけではないので注意が必要なものの、やはり物理化学的なパラメータだと従来型の機械学習よりもGNNが強い印象です。
次回のモデル構築編3回目は少し変わり種で、表形式データに特化したニューラルネットワークモデルを試したいと思います。
コンペティションで使われることがあるモデルという程度で、ケモインフォマティクスでの利用実績は(恐らく)報告されておらず、従来型のモデルとの差別化点が少ないこともあり正直現実では使い時があまりありません。
ですが、機械学習の世界は進歩が早いので、近い将来に発展形のモデルが従来型のモデルに取って代わる可能性もありえます。特性を頭に入れておいて損はないと思うので、次回のタイミングで紹介したいと思います。




コメント