創薬向けのOSSである「Pharmit」をローカル環境で動かしてみたいと思います。
実行環境
- OS:WSL2, Ubuntu 24.04 (Windows 11 Pro)
- CPU: Intel(R) Core(TM) Ultra 9 185H (WSL2プロセッサ数; 22コア)
- GPU: NVIDIA GeForce RTX 4070 (8GB)
- RAM: 32GB
Pharmitについて
Pharmitは、ファーマコフォアベースのバーチャルスクリーニングをサポートするOSSです。
ファーマコフォア関連はLigand-Based Drug Design (LBDD) に分類されますが、Pharmitでは標的タンパク質の結合ポケット内でのエネルギー最適化やドッキングスコアの算出といったStructure-Based Drug Design (SBDD)-likeな機能も搭載されています。
Pharmitは、Web上で公開されているアプリケーションが利用可能ですが、営利組織では秘密保持や権利化等の問題で用いるのは現実的ではありません。
今回は、勤め先でも利用できるようにローカル環境へとPharmitを導入してみたいと思います。また、実際にクエリ作成と類似度検索を行い操作感を試してみたいと思います。
※本記事では、ファーマコフォアを考慮した検索のみに触れます。結合ポケットを加味した排除体積の設定や結合ポケット内でのエネルギー最適化といったことは行いません。
環境構築
Githubを参考に、Ubuntu22.04向けのインストール方法を試します(手順はWSL2環境で成功したものです)。
Pharmitを動かすためには、①FastCGI 、②Open Babel、③smina、この3つのソフトウェアが必要です。
本記事では①③は利用しませんが、ビルドに必要となるので合わせて導入します。
まずは、ソフトウェアのビルドに必要なライブラリやアプリケーションをaptでインストールしましょう。
sudo apt install git autoconf automake libtool ghostscript liblemon-dev libeigen3-dev libann-dev bmagic libcgicc-dev libgoogle-perftools-dev libglpk-dev coinor-* libjsoncpp-dev cmake libboost-dev swig libxml2-dev libcairo2-dev libboost-all-dev libcurl4-openssl-devインストールが完了したら、ソフトウェアをインストールするためのディレクトリに移動しましょう。場所はどこでもよいですが、ユーザーディレクトリの直下にディレクトリを作成するのが無難だと思います。
cd (任意のディレクトリ)以下、各ソフトウェアをインストールしていきます。
FastCGI
FastCGIは、Webサーバーとアプリケーションサーバー間の通信プロトコルの一種です。
エンジニアへの転職を考えていない限り創薬の研究者は「これがあることでPharmitをWebアプリケーションで利用できる」くらいの認識で問題ないと思います。
以下のコマンドでインストールします。インストールの速度を上げたい場合は、makeコマンド実行時に”-j8”のように引数を渡すと数字に対応したCPU数を利用できます。
git clone https://github.com/FastCGI-Archives/fcgi2.git
cd fcgi2
autoreconf -i
./configure
sudo make
sudo make install
cd ..Open Babel
Open Babelは、化学構造の前処理を得意とするOSSです。結合次数のアサインがRDkitよりも正確なことが多く、リガンドのPDBファイルをSDFに変換する際によく使われている印象です。
インストールは以下のコマンドで実行します。先ほどと同様、インストールの速度を上げたい場合は、makeコマンド実行時に”-j8”のように引数を渡すと数字に対応したCPU数を利用できます。
git clone https://github.com/openbabel/openbabel.git
cd openbabel
mkdir build
cd build
sudo cmake .. -DPYTHON_BINDINGS=1 -DRUN_SWIG=1 -DWITH_MAEPARSER=0 -DWITH_COORDGEN=0
sudo make
sudo make install
cd ../..必須ではないですが、実行ファイルと関連ライブラリの保存先にパスを通しておくと便利です。方法は色々ありますが、私はユーザー直下の.bashrcに以下の行を追加しました(optは最初に移動したインストール先のディレクトリ)。
# openbabel-3.1.1
if [ -x "$HOME/opt/openbabel/build/bin" ]; then
export PATH="$HOME/opt/openbabel/build/bin:$PATH"
export BABEL_LIBDIR="$HOME/opt/openbabel/build/lib"
fiこれで次回以降に立ち上げたシェルではobabelコマンドが利用できます。現在のシェルでも実行したい場合は、以下のコマンドを実行しましょう。
source ~/.bashrcsmina
sminaは、ドッキングシミュレーション用のOSSです。AutoDock vinaというドッキングシミュレーション用OSSの派生版で、Phamitでの結合親和性スコアの算出に用いられています。
インストールは以下のコマンドで実行します。先ほどと同様、インストールの速度を上げたい場合は以下略。
git clone http://git.code.sf.net/p/smina/code smina
cd smina
mkdir build
cd build
sudo cmake .. -DCMAKE_CXX_FLAGS="-DBOOST_TIMER_ENABLE_DEPRECATED"
sudo make -j8
sudo cp libsmina.a /usr/local/lib/libsmina.aこちらも必須ではないですが、実行ファイルのパスを通しておくと便利です(以下、.bashrcの参考、optは最初に移動したインストール先のディレクトリ)。
# smina
if [ -x "$HOME/opt/smina/build/smina" ]; then
export PATH="$HOME/opt/smina/build:$PATH"
fiこれで次回以降に立ち上げたシェルではsminaコマンドが利用できます。
Parmit
最後に、Phatmitをインストールします。まずは、Githubからリポジトリをダウンロードしましょう。
git clone http://git.code.sf.net/p/pharmit/code pharmit次に、ビルド時のエラー回避用にファイルを作成します。
私がGithub記載のインストール方法を試した際、グラフ構造データの計算に利用する「LEMON」というライブラリ関係でエラーが発生しました。
ChatGPTに色々と質問したところ、ライブラリ自体はインストールされている一方で、ビルド時にうまくファイルを見つけられていないことが原因だとわかりました。
これを回避するために、FindLEMON.cmakeというファイルを(ChatGPTが)作成しました。まずは、以下の手順で所定の場所にファイルを作成します。
cd pharmit/src/cmake
touch FindLEMON.cmake作成したFindLEMON.cmakeに、以下の内容を記載して保存します。
# Minimal finder for Ubuntu's liblemon-dev
find_path(LEMON_INCLUDE_DIR NAMES lemon/list_graph.h PATHS /usr/include /usr/local/include)
find_library(LEMON_LIBRARY NAMES lemon liblemon PATHS /usr/lib /usr/lib/x86_64-linux-gnu /usr/local/lib)
include(FindPackageHandleStandardArgs)
find_package_handle_standard_args(LEMON DEFAULT_MSG LEMON_INCLUDE_DIR LEMON_LIBRARY)
if(LEMON_FOUND)
set(LEMON_INCLUDE_DIRS ${LEMON_INCLUDE_DIR})
set(LEMON_LIBRARIES ${LEMON_LIBRARY})
endif()
mark_as_advanced(LEMON_INCLUDE_DIR LEMON_LIBRARY)これで、ビルド時のエラー回避が完了しました。あとは、以下の手順でPharmitをビルドします。インストールの速度を上げたい場合は以下略。
cd ..
make build
cd build
cmake .. -DSMINALIB=/usr/local/lib/libsmina.a -DSMINA_DIR=$HOME/opt/smina/ -DLEMON_DIR=$HOME/opt/lemon-1.3.1/ -DCMAKE_CXX_FLAGS="-include cassert -DBOOST_TIMER_ENABLE_DEPRECATED"
make
sudo make install以上でPhartmitのインストールは完了です。
Pharmitの操作
今回はEGFRの阻害剤4種のうち、1種をクエリ、残りを検索対象としてバーチャルスクリーニングを行いたいと思います。
データ準備
Protein Data Bank (PDB) から、5HG8、5UG8、5UG9、5UGC、この4つのファイルを「Legacy PDB format」でダウンロードし、PyMOLでリガンドを抽出します。
PyMOLはconda環境にインストールして利用します。仮想環境の作成、インストール、起動の方法は以下の通りです。
conda create -n pymol python=3.10
conda activate pymol
conda install pymol-open-source
pymolPyMOLを起動したらダウンロードしたPDBファイルを読み込みます。
リガンドを抽出したいところですが、5HG8の座標の重心が他のファイルとずれています。
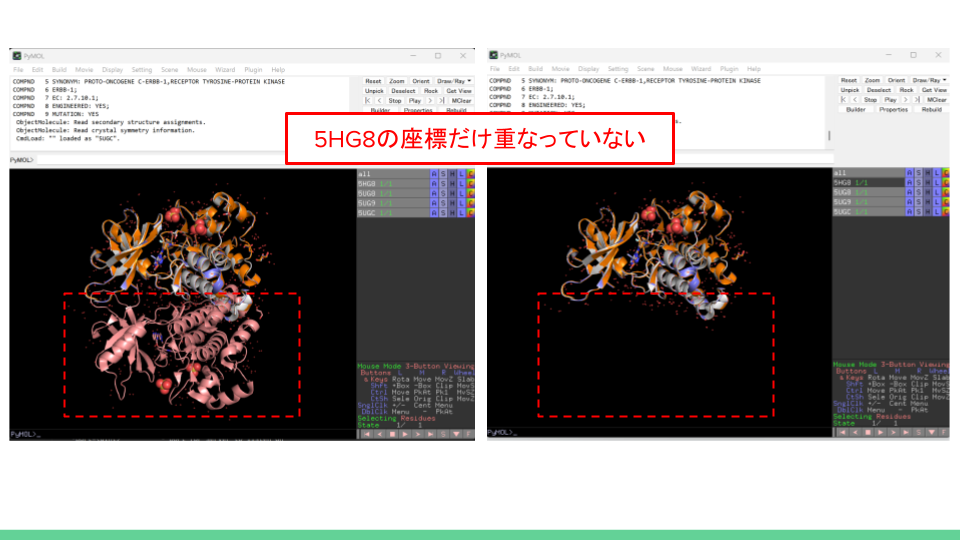
ファーマコフォアは三次元座標に依存するため、alignコマンドで5UG8の座標と揃えます。PyMOL上で以下のコマンドを実行しましょう。
align 5HG8, 5UG8これで座標がそろいました。次に、複合体からリガンドを抽出します。
リガンドの抽出方法は、GUI上でリガンドを選択 > (sele) の A > extract object で可能です。リガンドが抽出出来たら、①リガンドごとのSDF、②全てのリガンドを含むSDF、この2パターンで保存します。
保存したSDFはテスト用のディレクトリを作成して、そこに保存しましょう。
クエリ作成
4つのリガンドのうち、5HG8のみ不飽和環が飽和環となっているところがあります。今回のクエリ作成は5UGCを基準として行い、5HG8を除く2つがヒットするどうかを確認してみます。
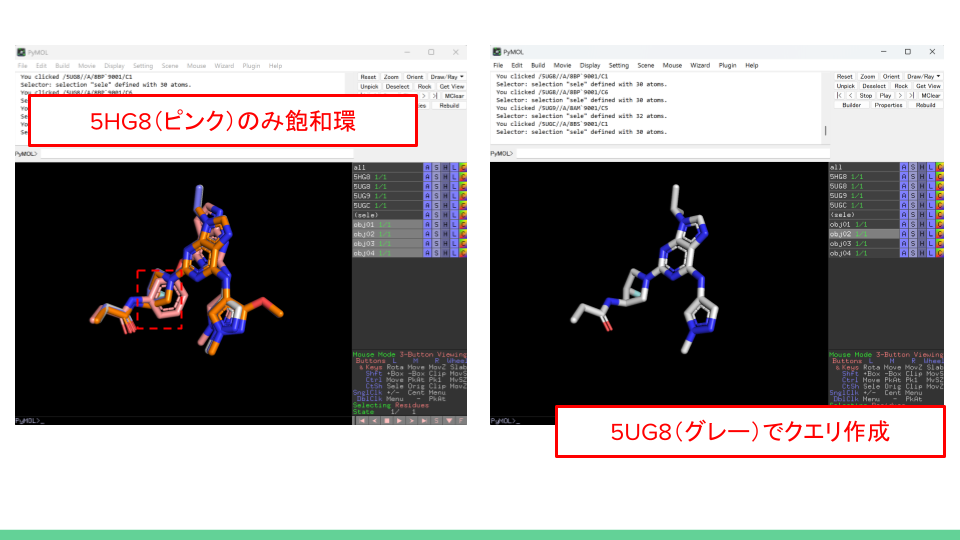
テスト用のディレクトリに移動して以下のコマンドを実行します。
cd (テスト用のディレクトリ)
pharmit -cmd pharma -in 5UG8.sdf -out PC_5UG8.jsonこれでテスト用のディレクトリに「PC_5UG8.json」というファイルができました。エラーが発生するかもしれませんが、今回のケースに関してはファイルができていれば問題ありません。
データベース作成
次に、検索対象のデータベースを作成します。実行は以下のコマンドです(merge.sdf は全てのリガンドを含むファイル名)。
pharmit -cmd dbcreate -dbdir DB -in merge.sdfこれでテスト用のディレクトリに「DB」というディレクトリが作成されました。こちらもエラーが発生するかもしれませんが、問題なく最後まで実行できました。
ファーマコフォア検索
最後に作成したクエリとデータベースを用いて検索を行います。コマンドは以下の通りです。
pharmit -cmd dbsearch -dbdir ./DB -in PC_5UG8.json -out result.sdfこれでテスト用のディレクトリに「result.sdf」というファイルが作成されました。
検索の結果、ヒットしたのは3つでした。ヒットしたのはクエリ+5HG8を除く2つのリガンドだったので、意図したような結果が得られたことになります。
最後に
創薬向けのOSSである「Pharmit」をローカル環境で動かしてみました。
今回クエリはデフォルトのまま利用しましたが、jsonファイルを編集することで特定のファーマコフォアのみに限定したクエリも作成可能なようです。
個人的な印象として、ファーマコフォア関連は商用ソフトウェアのほうがやはり便利です。
一方で、商用ソフトの場合、ライセンスやトークンが競合して使いたいときに使えない可能性があったり、アプリケーション間の連携にひと手間必要だったりします(生成AIの報酬に利用したい場合など)。
そういった意味では、1つくらいファーマコフォア関連のOSSを扱えるようにしておいても良いと思いました。
商用ソフトは独学に使いづらいということもあるので、ファーマコフォア関連も引き続きOSSを追っていきたいと思います。




コメント